Indledning
Beslutningstræer er en type superviseret læringsalgoritme, der kan bruges til både regressions- og klassifikationsproblemer. Den fungerer for både kategoriske og kontinuerlige input- og outputvariabler.
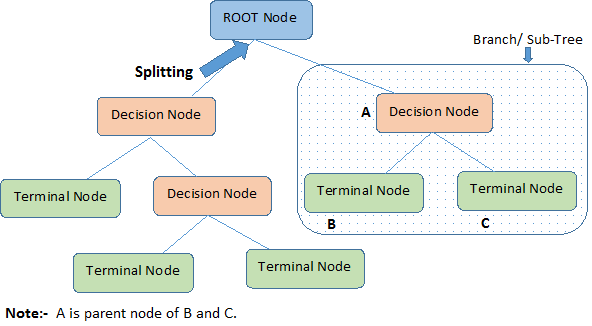
Lad os identificere vigtige terminologier om beslutningstræ, idet vi ser på billedet ovenfor:
-
Root Node repræsenterer hele populationen eller prøven. Den bliver yderligere opdelt i to eller flere homogene sæt.
-
Splitting er en proces, hvor en knude deles op i to eller flere underknuder.
-
Når en underknude deles op i yderligere underknuder, kaldes den en beslutningsknude.
-
Noder, der ikke deler sig, kaldes en terminalknude eller et blad.
-
Når man fjerner underknuder af en beslutningsknude, kaldes denne proces for beskæring. Det modsatte af beskæring er Splitting.
-
En underafdeling af et helt træ kaldes Branch.
-
En knude, som er opdelt i underknuder, kaldes en forældreknude til underknuderne; mens underknuderne kaldes barn af forældreknuden.
Typer af beslutningstræer
Regressionstræer
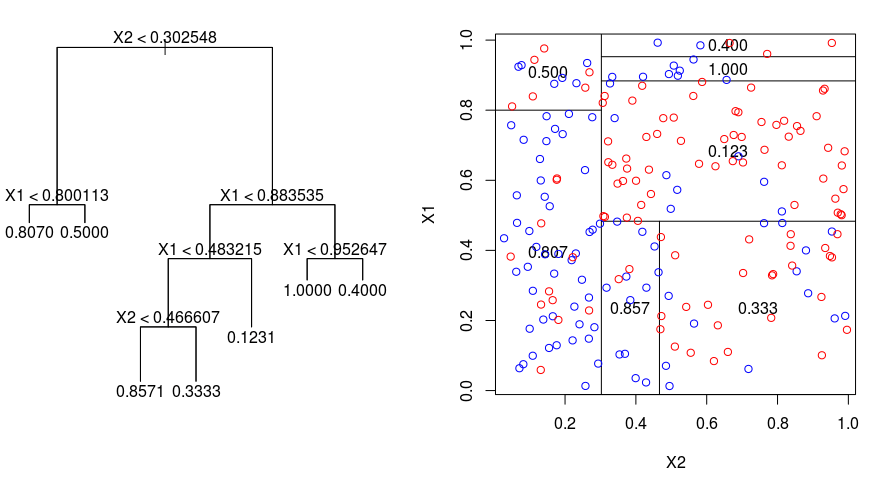
Lad os tage et kig på nedenstående billede, som hjælper med at visualisere arten af den opdeling, der udføres af et regressionstræ. Dette viser et ubeskåret træ og et regressionstræ, der passer til et tilfældigt datasæt. Begge visualiseringer viser en række opdelingsregler, der starter i toppen af træet. Bemærk, at hver opdeling af domænet er afstemt med en af funktionsakserne. Begrebet parallel opsplitning af akser kan uden videre generaliseres til dimensioner større end to. For et funktionsrum af størrelse $p$, en delmængde af $\mathbb{R}^p$, opdeles rummet i $M$-regioner, $R_{m}$, som hver især er en $p$-dimensionel “hyperblok”.
For at opbygge et regressionstræ bruger man først rekursiv binær splititng til at vokse et stort træ på træningsdataene og stopper først, når hver terminalknude har færre end et vist minimumsantal observationer. Rekursiv binær opsplitning er en grådig og top-down-algoritme, der anvendes til at minimere residualsummen af kvadrater (RSS), et fejlmål, der også anvendes i lineære regressionsindstillinger. RSS, i tilfælde af et partitioneret feature space med M partitioner er givet ved:
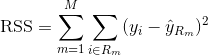
Med udgangspunkt i toppen af træet deler man det op i 2 grene, hvorved der skabes en partition af 2 rum. Derefter udfører man denne særlige opdeling i toppen af træet flere gange og vælger den opdeling af funktionerne, der minimerer den (aktuelle) RSS.
Dernæst anvender man cost complexity pruning på det store træ for at opnå en sekvens af de bedste undertræer, som en funktion af $\alpha$. Den grundlæggende idé her er at indføre en ekstra tuningparameter, betegnet $\alpha$, der afbalancerer træets dybde og dets tilpasningsevne til træningsdataene.
Du kan bruge K-fold krydsvalidering til at vælge $\alpha$. Denne teknik indebærer simpelthen, at træningsobservationerne opdeles i K foldninger for at estimere testfejlprocenten for undertræerne. Dit mål er at vælge den, der fører til den laveste fejlprocent.
Klassifikationstræer
Et klassifikationstræ ligner meget et regressionstræ, bortset fra at det bruges til at forudsige et kvalitativt svar i stedet for et kvantitativt svar.
Husk, at for et regressionstræ er det forudsagte svar for en observation givet ved det gennemsnitlige svar for de træningsobservationer, der tilhører den samme terminale knude. I modsætning hertil forudsiger man for et klassifikationstræ, at hver observation tilhører den hyppigst forekommende klasse af træningsobservationer i den region, som den tilhører.
I fortolkningen af resultaterne af et klassifikationstræ er man ofte ikke kun interesseret i den klasseprædiktion, der svarer til en bestemt terminalknude-region, men også i klasseproportionerne blandt de træningsobservationer, der falder ind i denne region.
Opgaven med at dyrke et klassifikationstræ ligner meget opgaven med at dyrke et regressionstræ. Ligesom i regressionssituationen bruger man rekursiv binær opdeling til at dyrke et klassifikationstræ. I klassifikationsindstillingen kan residual sum of squares imidlertid ikke anvendes som kriterium for at foretage de binære opdelinger. I stedet kan du bruge en af disse 3 metoder nedenfor:
- Klassifikationsfejlprocent: I stedet for at se, hvor langt et numerisk svar er væk fra middelværdien, som i regressionsindstillingen, kan du i stedet definere “hit rate” som den brøkdel af træningsobservationer i et bestemt område, der ikke tilhører den mest udbredte klasse. Fejlen er givet ved denne ligning:
E = 1 – argmaxc($\hat{\pi}_{mc}$)
hvor $\hat{\pi}_{mc}$ repræsenterer den brøkdel af træningsdata i region Rm, der tilhører klasse c.
- Gini-indeks: Gini-indekset er en alternativ fejlmetrik, der er beregnet til at vise, hvor “ren” en region er. “Renhed” betyder i dette tilfælde, hvor stor en del af træningsdataene i en bestemt region der tilhører en enkelt klasse. Hvis en region Rm indeholder data, der hovedsagelig stammer fra en enkelt klasse c, vil Gini-indeksværdien være lille:
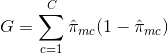
- Krydsentropi: Et tredje alternativ, som ligner Gini-indekset, er kendt som krydsentropi eller afvigelse:
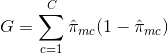
Krydsentropien vil antage en værdi nær nul, hvis $\hat{\pi}_{mc}$’erne alle er nær 0 eller nær 1. Derfor vil krydsentropien, ligesom Gini-indekset, antage en lille værdi, hvis den m-te knude er ren. Det viser sig faktisk, at Gini-indekset og krydsentropien numerisk set er ret ens.
Ved opbygning af et klassifikationstræ anvendes enten Gini-indekset eller krydsentropien typisk til at vurdere kvaliteten af en bestemt opdeling, da de er mere følsomme over for renhed af knuder end klassifikationsfejlprocenten. Enhver af disse 3 fremgangsmåder kan anvendes ved beskæring af træet, men klassifikationsfejlprocenten er at foretrække, hvis forudsigelsesnøjagtigheden af det endelige beskårede træ er målet.
For- og ulemper ved beslutningstræer
Den største fordel ved at anvende beslutningstræer er, at de intuitivt set er meget lette at forklare. De afspejler nøje den menneskelige beslutningstagning sammenlignet med andre regressions- og klassifikationsmetoder. De kan vises grafisk, og de kan nemt håndtere kvalitative prædiktorer uden at skulle oprette dummy-variabler.
Derimod har beslutningstræer generelt ikke den samme grad af forudsigelsesnøjagtighed som andre tilgange, da de ikke er helt robuste. En lille ændring i dataene kan medføre en stor ændring i det endelige estimerede træ.
Gennem aggregering af mange beslutningstræer ved hjælp af metoder som bagging, randomforests og boosting kan beslutningstræernes forudsigelsespræstation forbedres væsentligt.
Træbaserede metoder
Bagging
De beslutningstræer, der er omtalt ovenfor, lider under høj varians, hvilket betyder, at hvis du deler træningsdataene tilfældigt op i to dele og tilpasser et beslutningstræ til begge halvdele, kan de resultater, du får, være meget forskellige. I modsætning hertil vil en procedure med lav varians give lignende resultater, hvis den anvendes gentagne gange på forskellige datasæt.
Bagging, eller bootstrap-aggregation, er en teknik, der bruges til at reducere variansen af dine forudsigelser ved at kombinere resultatet af flere klassifikatorer, der er modelleret på forskellige delprøver af det samme datasæt. Her er ligningen for bagging:

hvor du genererer $B$ forskellige bootstrappede træningsdatasæt. Derefter træner du din metode på det $bth$ bootstrappede træningssæt for at få $\hat{f}_{b}(x)$ og til sidst beregner du gennemsnittet af forudsigelserne.
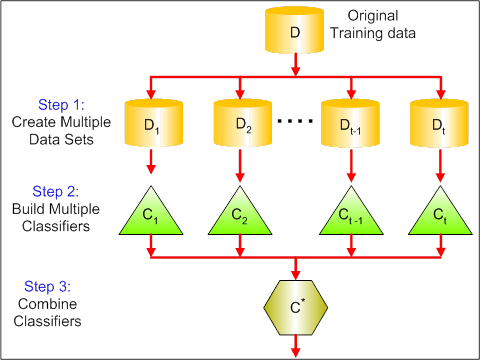
Det visuelle billede nedenfor viser de 3 forskellige trin i bagging:
-
Strin 1: Her erstatter du de oprindelige data med nye data. De nye data har som regel en brøkdel af de oprindelige datas kolonner og rækker, som så kan bruges som hyperparametre i bagging-modellen.
-
Stræk 2: Du opbygger klassifikatorer på hvert datasæt. Generelt kan du bruge den samme klassifikator til at lave modeller og forudsigelser.
-
Stræk 3: Til sidst bruger du en gennemsnitsværdi til at kombinere forudsigelserne fra alle klassifikatorer, afhængigt af problemet. Generelt er disse kombinerede værdier mere robuste end en enkelt model.
Som bagging kan forbedre forudsigelserne for mange regressions- og klassifikationsmetoder, er det især nyttigt for beslutningstræer. For at anvende bagging på regressions-/klassifikationstræer konstruerer man simpelthen $B$ regressions-/klassifikationstræer ved hjælp af $B$ bootstrappede træningssæt og beregner gennemsnittet af de resulterende forudsigelser. Disse træer vokser dybt, og de beskæres ikke. Derfor har hvert enkelt træ en høj varians, men en lav bias. Ved at beregne gennemsnittet af disse $B$ træer reduceres variansen.
Bredt sagt har bagging vist sig at give imponerende forbedringer i nøjagtighed ved at kombinere hundredvis eller endog tusindvis af træer i en enkelt procedure.
Random Forests
Random Forests er en alsidig maskinlæringsmetode, der kan udføre både regressions- og klassifikationsopgaver. Den foretager også dimensionelle reduktionsmetoder, behandler manglende værdier, outlier-værdier og andre væsentlige trin i dataudforskningen og gør et ret godt stykke arbejde.
Random Forests giver en forbedring i forhold til bagged trees ved hjælp af en lille justering, der dekorrelerer træerne. Som i bagging opbygger du et antal beslutningstræer på bootstrappede træningsprøver. Men ved opbygningen af disse beslutningstræer vælges der, hver gang der overvejes en opdeling i et træ, en tilfældig stikprøve af m prædiktorer som opdelingskandidater fra det fulde sæt af $p$ prædiktorer. Opdelingen må kun bruge én af disse $m$ prædiktorer. Dette er den væsentligste forskel mellem tilfældige skove og bagging; for ligesom i bagging er valget af prædiktor $m = p$.
For at dyrke en tilfældig skov skal du:
-
Først antages det, at antallet af tilfælde i træningsmængden er K. Derefter skal du tage en tilfældig stikprøve af disse K tilfælde og derefter bruge denne stikprøve som træningssæt til at dyrke træet.
-
Hvis der er $p$ inputvariabler, skal du angive et antal $m < p$, således at du ved hver knude kan vælge $m$ tilfældige variabler ud af $p$. Den bedste opdeling på disse $m$ bruges til at opdele knuden.
-
Hvert træ vokser efterfølgende i størst muligt omfang, og der er ikke behov for beskæring.
-
Slutteligt aggregeres forudsigelserne fra måltræerne for at forudsige nye data.
Random Forests er meget effektiv til at estimere manglende data og opretholde nøjagtigheden, når en stor andel af dataene mangler. Den kan også afbalancere fejl i datasæt, hvor klasserne er ubalancerede. Det vigtigste er, at den kan håndtere massive datasæt med stor dimensionalitet. En ulempe ved at bruge Random Forests er dog, at man let kan overpasse støjende datasæt, især når man foretager regression.
Boosting
Boosting er en anden metode til at forbedre de forudsigelser, der er resultatet af et beslutningstræ. Ligesom bagging og randomforests er det en generel tilgang, der kan anvendes på mange statistiske indlæringsmetoder til regression eller klassificering. Husk, at bagging indebærer, at der oprettes flere kopier af det oprindelige træningsdatasæt ved hjælp af bootstrap, at der tilpasses et separat beslutningstræ til hver kopi, og at alle træerne derefter kombineres for at skabe en enkelt forudsigelsesmodel. Det skal bemærkes, at hvert træ er opbygget på et bootstrapped datasæt, uafhængigt af de andre træer.
Boosting fungerer på samme måde, bortset fra at træerne dyrkes sekventielt: hvert træ dyrkes ved hjælp af oplysninger fra tidligere dyrkede træer. Boosting indebærer ikke bootstrap-prøvetagning; i stedet tilpasses hvert træ på en modificeret version af det oprindelige datasæt.

For både regressions- og klassifikationstræer fungerer boosting således:
-
I modsætning til at tilpasse et enkelt stort beslutningstræ til dataene, hvilket svarer til at tilpasse dataene hårdt og potentielt overfitte, lærer boosting-tilgangen i stedet langsomt.
-
Givet den aktuelle model tilpasser du et beslutningstræ til residualerne fra modellen. Det vil sige, at du tilpasser et træ ved hjælp af de aktuelle residualer, snarere end resultatet $Y$, som svar.
-
Du tilføjer derefter dette nye beslutningstræ til den tilpassede funktion for at opdatere residualerne. Hvert af disse træer kan være ret lille, med kun nogle få terminale knuder, som bestemmes af parameteren $d$ i algoritmen. Ved at tilpasse små træer til residualerne forbedrer du langsomt $\hat{f}$ på områder, hvor den ikke klarer sig godt.
-
Krympningsparameteren $\nu$ bremser processen yderligere, så flere og anderledes formede træer kan angribe residualerne.
Boosting er meget nyttigt, når du har mange data, og du forventer, at beslutningstræerne bliver meget komplekse. Boosting er blevet brugt til at løse mange udfordrende klassifikations- og regressionsproblemer, herunder risikoanalyse, følelsesanalyse, prædiktiv reklame, prismodellering, salgsestimat og patientdiagnose, blandt andet.
Decision Trees in R
Classification Trees
I denne del arbejder du med datasættet Carseats ved hjælp af pakken tree i R. Vær opmærksom på, at du først skal installere pakkerne ISLR og tree i dit R Studio-miljø. Lad os først indlæse Carseats-datasættet fra ISLR-pakken.
library(ISLR)data(package="ISLR")carseats<-CarseatsLad os også indlæse tree-pakken.
require(tree)Datasættet Carseats er et dataframe med 400 observationer på følgende 11 variabler:
-
Salg: Salg pr. enhed i tusinde
-
Kompris: pris, der opkræves af konkurrenten på hvert sted
-
Indkomst: indkomstniveau i lokalsamfundet i tusindvis af dollars
-
Reklame: lokalt annoncebudget på hvert sted i tusindvis af dollars
-
Befolkning: regional befolkning i tusindvis
-
Pris: pris for autostole på hvert sted
-
ShelveLoc: Bad, Good eller Medium angiver kvaliteten af hyldeplacering
-
Age: aldersniveau for befolkningen
-
Education: ed niveau på stedet
-
Urban: Ja/Nej
-
USA: Ja/Nej
names(carseats)Lad os tage et kig på histogrammet over bilsalg:
hist(carseats$Sales)Opmærksomheden henledes på, at Sales er en kvantitativ variabel. Du vil gerne demonstrere den ved hjælp af træer med et binært svar. For at gøre det, omdanner du Sales til en binær variabel, som vil blive kaldt High. Hvis salget er mindre end 8, vil det ikke være højt. I modsat fald vil den være høj. Derefter kan du sætte den nye variabel High tilbage i dataframe.
High = ifelse(carseats$Sales<=8, "No", "Yes")carseats = data.frame(carseats, High)Nu skal vi udfylde en model ved hjælp af beslutningstræer. Du kan naturligvis ikke have Sales-variablen her, fordi din responsvariabel High blev oprettet ud fra Sales. Lad os derfor udelukke den og tilpasse træet.
tree.carseats = tree(High~.-Sales, data=carseats)Lad os se oversigten over dit klassifikationstræ:
summary(tree.carseats)Du kan se de involverede variabler, antallet af terminale knuder, den resterende gennemsnitlige afvigelse samt fejlprocenten for fejlklassificering. For at gøre det mere visuelt, lad os også plotte træet og derefter annotere det ved hjælp af den praktiske text-funktion:
plot(tree.carseats)text(tree.carseats, pretty = 0)Der er så mange variabler, hvilket gør det meget kompliceret at se på træet. I det mindste kan du se, at ved hver af de terminale knuder er de mærket med Yes eller No. Ved hver opdelingsknude vises variablerne og værdien af opdelingsvalget (f.eks. Price < 92.5 eller Advertising < 13.5).
For at få et detaljeret resumé af træet skal du blot udskrive det. Det vil være praktisk, hvis du ønsker at udtrække detaljer fra træet til andre formål:
tree.carseatsDet er tid til at beskære træet ned. Lad os oprette et træningssæt og et testsæt ved at opdele datarammen carseats i 250 træningsprøver og 150 testprøver. Først indstiller du et frø for at gøre resultaterne reproducerbare. Derefter tager du et tilfældigt udsnit af ID-numrene (indeksnumrene) for prøverne. Specifikt her udtager du en prøve fra sættet 1 til n række antal rækker af bilsæder, som er 400. Du ønsker en stikprøve af størrelse 250 (som standard bruger stikprøven uden udskiftning).
set.seed(101)train=sample(1:nrow(carseats), 250)Så nu får du dette indeks på train, som indekserer 250 af de 400 observationer. Du kan genindstille modellen med tree ved hjælp af den samme formel, bortset fra at du fortæller træet, at det skal bruge en delmængde, der er lig med train. Lad os så lave et plot:
tree.carseats = tree(High~.-Sales, carseats, subset=train)plot(tree.carseats)text(tree.carseats, pretty=0)Plottet ser en smule anderledes ud på grund af det lidt anderledes datasæt. Ikke desto mindre ser træets kompleksitet nogenlunde den samme ud.
Nu skal du tage dette træ og forudsige det på testmængden ved hjælp af predict-metoden for træer. Her skal du faktisk forudsige class-etiketterne.
tree.pred = predict(tree.carseats, carseats, type="class")Dernæst kan du evaluere fejlen ved hjælp af en fejlklassifikationstabel.
with(carseats, table(tree.pred, High))På diagonalerne er de korrekte klassifikationer, mens uden for diagonalerne er de forkerte klassifikationer. Du ønsker kun at geninddele de korrekte. For at gøre det kan du tage summen af de 2 diagonaler divideret med det samlede antal (150 testobservationer).
(72 + 43) / 150Ok, du får en fejl på 0,76 med dette træ.
Når man dyrker et stort busket træ, kan det have for stor varians. Lad os derfor bruge krydsvalidering til at beskære træet optimalt. Ved hjælp af cv.tree bruger du fejlklassifikationsfejlen som grundlag for at foretage beskæringen.
cv.carseats = cv.tree(tree.carseats, FUN = prune.misclass)cv.carseatsUdskrivningen af resultaterne viser detaljerne om stien for krydsvalidering. Du kan se størrelsen af træerne, efterhånden som de blev beskåret, afvigelserne, efterhånden som beskæringen fortsatte, samt den omkostningskompleksitetsparameter, der blev anvendt i processen.
Lad os plotte dette ud:
plot(cv.carseats)Kigger du på plottet, ser du en nedadgående spiraldel på grund af fejlklassifikationsfejlen på 250 krydsvaliderede punkter. Så lad os vælge en værdi i de nedadgående trin (12). Lad os derefter beskære træet til en størrelse på 12 for at identificere dette træ. Lad os endelig plotte og annotere dette træ for at se resultatet.
prune.carseats = prune.misclass(tree.carseats, best = 12)plot(prune.carseats)text(prune.carseats, pretty=0)Det er en smule fladere end tidligere træer, og man kan faktisk læse etiketterne. Lad os evaluere det på testdatasættet igen.
tree.pred = predict(prune.carseats, carseats, type="class")with(carseats, table(tree.pred, High))(74 + 39) / 150Det ser ud til, at de korrekte klassifikationer er faldet en lille smule. Det har gjort omtrent det samme som dit oprindelige træ, så beskæringen gjorde ikke meget skade med hensyn til fejlklassifikationsfejl og gav et enklere træ.
Ofte giver træer ikke særlig gode forudsigelsesfejl, så lad os tage et kig på randomforests og boosting, som har en tendens til at overgå træer med hensyn til forudsigelse og fejlklassificering.
Randomforests
I denne del vil du bruge Boston housing data til at udforske randomforests og boosting. Datasættet er placeret i pakken MASS. Det giver boligværdier og andre statistikker i hver af 506 forstæder i Boston baseret på en folketælling fra 1970.
library(MASS)data(package="MASS")boston<-Bostondim(boston)names(boston)Lad os også indlæse randomForest-pakken.
require(randomForest)For at forberede data til random forest skal vi indstille seed og oprette et prøve-træningssæt på 300 observationer.
set.seed(101)train = sample(1:nrow(boston), 300)I dette datasæt er der 506 forstæder i Boston. For hver surburb har du variabler som f.eks. kriminalitet pr. indbygger, typer af industri, gennemsnitligt antal værelser pr. bolig, gennemsnitlig andel af husenes alder osv. Lad os bruge medv – medianværdien af ejerboliger for hver af disse surburbs – som responsvariabel.
Lad os tilpasse en random forest og se, hvor godt den klarer sig. Som sagt bruger du svaret medv, medianværdien af boliger (i 1.000 dollars) og træningsprøvesættet.
rf.boston = randomForest(medv~., data = boston, subset = train)rf.bostonUdskrivningen af den tilfældige skov giver en oversigt over den: antal træer (der blev dyrket 500), de gennemsnitlige kvadrerede residualer (MSR) og den procentdel af variansen, der forklares. MSR og % varians forklaret er baseret på out-of-bag-estimaterne, en meget smart anordning i randomforests til at få ærlige fejlestimater.
Den eneste tuningparameter i en random Forests er argumentet kaldet mtry, som er antallet af variabler, der vælges ved hvert split af hvert træ, når du laver et split. Som det ses her, er mtry 4 af de 13 udforskende variabler (eksklusive medv) i Boston Housing-dataene – hvilket betyder, at hver gang træet kommer til at splitte en knude, vil 4 variabler blive udvalgt tilfældigt, hvorefter splittet vil blive begrænset til 1 af disse 4 variabler. Det er sådan randomForests dekorrelerer træerne.
Du vil tilpasse en række tilfældige skove. Der er 13 variabler, så lad os lade mtry spænde fra 1 til 13:
-
For at registrere fejlene opstiller du 2 variabler
oob.errogtest.err. -
I en løkke af
mtryfra 1 til 13 tilpasser du førstrandomForestmed denne værdi afmtrypå datasættettrain, idet du begrænser antallet af træer til at være 350. -
Dernæst uddrager du den gennemsnitlige kvadratiske fejl på objektet (out-of-bag-fejl).
-
Dernæst forudsiger du på testdatasættet (
boston) ved hjælp affit(tilpasningen afrandomForest). -
Sidst beregner du testfejlen: den gennemsnitlige kvadratiske fejl, som er lig med
mean( (medv - pred) ^ 2 ).
oob.err = double(13)test.err = double(13)for(mtry in 1:13){ fit = randomForest(medv~., data = boston, subset=train, mtry=mtry, ntree = 350) oob.err = fit$mse pred = predict(fit, boston) test.err = with(boston, mean( (medv-pred)^2 ))}Sammenfattende har du lige dyrket 4550 træer (13 gange 350). Lad os nu lave et plot ved hjælp af kommandoen matplot. Testfejlen og out-of-bag-fejlen bindes sammen til en 2-kolonne-matrix. Der er et par andre argumenter i matricen, herunder værdierne for plottetegn (pch = 23 betyder fyldt diamant), farver (rød og blå), type er lig med begge (plotter begge punkter og forbinder dem med linjerne) og navnet på y-aksen (Mean Squared Error). Du kan også sætte en legende øverst i højre hjørne af plottet.
matplot(1:mtry, cbind(test.err, oob.err), pch = 23, col = c("red", "blue"), type = "b", ylab="Mean Squared Error")legend("topright", legend = c("OOB", "Test"), pch = 23, col = c("red", "blue"))Disse 2 kurver burde egentlig være på linje, men det ser ud til, at testfejlen er en smule lavere. Der er dog en stor variabilitet i disse testfejlskøn. Da out-of-bag fejlskønnet blev beregnet på et datasæt og testfejlskønnet blev beregnet på et andet datasæt, ligger disse forskelle ret godt inden for standardfejlene.
Mærker du, at den røde kurve ligger jævnt over den blå kurve? Disse fejlskøn er meget korrelerede, fordi randomForest med mtry = 4 ligner meget den med mtry = 5. Det er derfor, at hver af kurverne er ret glatte. Det, du ser, er, at mtry omkring 4 synes at være det mest optimale valg, i hvert fald for testfejlen. Denne værdi af mtry for out-of-bag-fejlen er lig med 9.
Så med meget få niveauer har du tilpasset en meget kraftig forudsigelsesmodel ved hjælp af randomforests. Hvordan det? Venstre side viser præstationen for et enkelt træ. Den gennemsnitlige kvadrerede fejl på out-of-bag er 26, og du er faldet ned til ca. 15 (lige lidt over halvdelen). Det betyder, at du har reduceret fejlen med halvdelen. På samme måde for testfejlen har du reduceret fejlen fra 20 til 12.
Boosting
Sammenlignet med tilfældige skove vokser boosting mindre og stubbier træer og går på skævheden. Du skal bruge pakken GBM (Gradient Boosted Modeling), i R.
require(gbm)GBM beder om fordelingen, som er Gaussisk, fordi du vil lave tab af kvadreret fejl. Du vil bede GBM om 10.000 træer, hvilket lyder som meget, men det vil være lavvandede træer. Interaktionsdybden er antallet af opdelinger, så du ønsker 4 opdelinger i hvert træ. Shrinkage er 0,01, hvilket er hvor meget du vil skrumpe træets trin tilbage.
boost.boston = gbm(medv~., data = boston, distribution = "gaussian", n.trees = 10000, shrinkage = 0.01, interaction.depth = 4)summary(boost.boston)Funktionen summary giver et plot med variabel vigtighed. Det ser ud til, at der er 2 variabler, der har høj relativ betydning: rm (antal værelser) og lstat (procentdel af personer med lavere økonomisk status i samfundet). Lad os plotte disse 2 variabler:
plot(boost.boston,i="lstat")plot(boost.boston,i="rm")Det 1. plot viser, at jo højere andelen af personer med lavere økonomisk status i forstaden er, jo lavere er værdien af boligpriserne. Det 2. plot viser den omvendte sammenhæng med antallet af værelser: det gennemsnitlige antal værelser i boligen stiger, når prisen stiger.
Det er tid til at forudsige en boosted model på testdatasættet. Lad os se på testpræstationen som en funktion af antallet af træer:
-
Først laver du et gitter af antallet af træer i trin på 100 fra 100 til 10.000.
-
Dernæst kører du
predict-funktionen på den boostede model. Den tagern.treessom argument og producerer en matrix af forudsigelser på testdataene. -
Dimensionerne i matrixen er 206 testobservationer og 100 forskellige forudsigelsesvektorer ved de 100 forskellige værdier af træer.
n.trees = seq(from = 100, to = 10000, by = 100)predmat = predict(boost.boston, newdata = boston, n.trees = n.trees)dim(predmat)
Det er tid til at beregne testfejlen for hver af de forudsigelsesvektorer:
-
predmater en matrix,medver en vektor, således er (predmat–medv) en matrix af forskelle. Du kan bruge funktionenapplytil kolonnerne for disse kvadratiske forskelle (middelværdien). Det ville beregne den kolonnevise gennemsnitlige kvadrerede fejl for de forudsigelsesvektorer. -
Dernæst laver du et plot ved hjælp af lignende parametre som det, der bruges til Random Forest. Det ville vise et plot med boosting error.
boost.err = with(boston, apply( (predmat - medv)^2, 2, mean) )plot(n.trees, boost.err, pch = 23, ylab = "Mean Squared Error", xlab = "# Trees", main = "Boosting Test Error")abline(h = min(test.err), col = "red")
Den boosting error falder temmelig meget i takt med, at antallet af træer stiger. Dette er et bevis på, at boosting ikke er tilbøjelig til at overpasse. Lad os også medtage den bedste testfejl fra randomForest i diagrammet. Boosting får faktisk et rimeligt beløb under testfejlen for randomForest.
Konklusion
Så det er slutningen på denne R-tutorial om opbygning af beslutningstræ-modeller: klassifikationstræer, randomforests og boostede træer. De 2 sidstnævnte er kraftfulde metoder, som du kan bruge når som helst efter behov. Min erfaring er, at boosting normalt udkonkurrerer RandomForest, men RandomForest er nemmere at implementere. I RandomForest er den eneste tuningparameter antallet af træer; mens der i boosting kræves flere tuningparametre ud over antallet af træer, herunder krympning og interaktionsdybde.
Hvis du gerne vil lære mere, skal du sørge for at tage et kig på vores kursus i Machine Learning Toolbox for R.