Introdução
Árvore de decisão é um tipo de algoritmo de aprendizagem supervisionada que pode ser usado tanto em problemas de regressão como de classificação. Ele funciona tanto para variáveis de entrada e saída categóricas como contínuas.
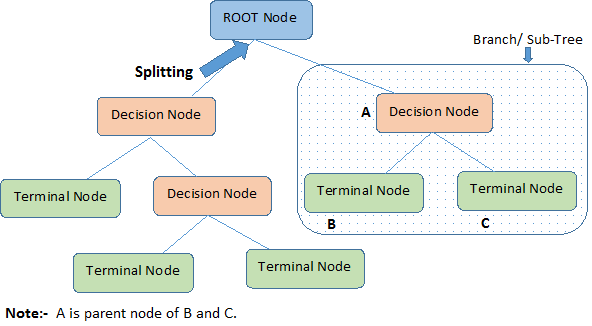
Deixe identificar terminologias importantes na árvore de decisão, olhando a imagem acima:
-
Nó Raiz representa toda a população ou amostra. Além disso, é dividido em dois ou mais conjuntos homogêneos.
-
Dividir é um processo de divisão de um nó em dois ou mais sub-nós.
-
Quando um sub-nó se divide em outros sub-nós, é chamado de Nó de Decisão.
-
Quando um sub-nó não se divide é chamado de Nó Terminal ou Folha.
-
Quando você remove sub-nós de um nó de decisão, este processo é chamado de Poda. O oposto da poda é a Poda dividida.
-
Uma sub-secção de uma árvore inteira é chamada de Ramo.
-
Um nó, que é dividido em sub-nós é chamado de nó pai dos sub-nós; enquanto os sub-nós são chamados de filho do nó pai.
Tipos de Árvores de Decisão
Árvores de Regressão
Vejamos a imagem abaixo, que ajuda a visualizar a natureza da partição feita por uma Árvore de Regressão. Isto mostra uma árvore não podada e uma árvore de regressão que se ajusta a um conjunto de dados aleatórios. Ambas as visualizações mostram uma série de regras de partição, começando no topo da árvore. Note que cada divisão do domínio está alinhada com um dos eixos de características. O conceito de divisão paralela de eixos generaliza-se de forma direta para dimensões superiores a dois. Para um espaço de características de tamanho $p$, um subconjunto de $\mathbb{R}^p$, o espaço é dividido em regiões $M$, $R_{m}$, cada uma das quais é um “hiperbloco” $p$-dimensional.
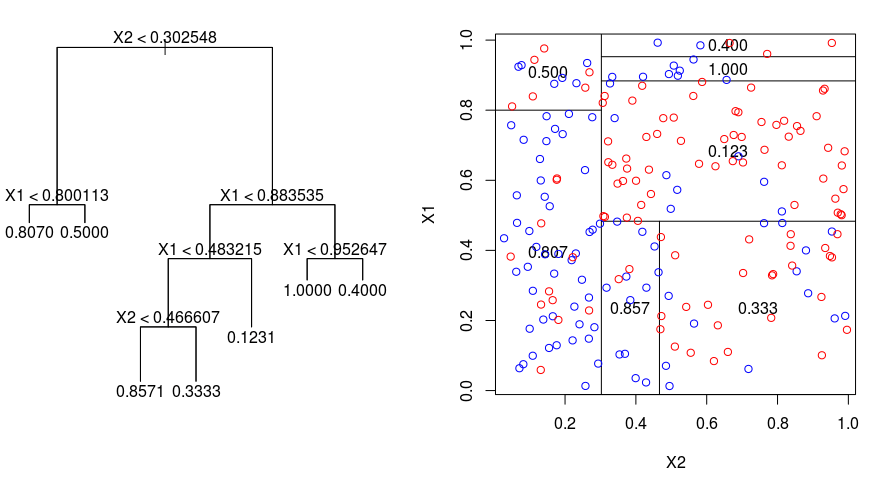
Para construir uma árvore de regressão, você primeiro usa splititng binário recursivo para fazer crescer uma árvore grande nos dados de treinamento, parando apenas quando cada nó terminal tem menos do que um número mínimo de observações. A Repartição Binária Recursiva é um algoritmo ganancioso e top-down usado para minimizar a Soma Residual dos Quadrados (RSS), uma medida de erro também usada em configurações de regressão linear. O RSS, no caso de um espaço particionado com partições M é dado por:
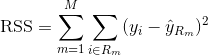 >
>
Beginning at the top of the tree, você o divide em 2 ramos, criando uma partição de 2 espaços. Em seguida, você executa essa partição particular no topo da árvore várias vezes e escolhe a divisão das características que minimiza o (atual) RSS.
Próximo, você aplica a poda de complexidade de custos à árvore grande, a fim de obter uma seqüência de melhores subárvores, em função de $\alpha$. A idéia básica aqui é introduzir um parâmetro de ajuste adicional, denotado por $\alpha$ que equilibra a profundidade da árvore e sua bondade de ajuste aos dados de treinamento.
Pode usar a validação cruzada de dobra K para escolher $\alpha$. Esta técnica envolve simplesmente a divisão das observações de treinamento em dobras K para estimar a taxa de erro do teste das sub-árvores. Seu objetivo é selecionar aquela que leva à menor taxa de erro.
Árvores de classificação
Uma árvore de classificação é muito semelhante a uma árvore de regressão, exceto que ela é usada para prever uma resposta qualitativa ao invés de uma quantitativa.
Recordar que para uma árvore de regressão, a resposta prevista para uma observação é dada pela resposta média das observações de treinamento que pertencem ao mesmo nó terminal. Em contraste, para uma árvore de classificação, você prevê que cada observação pertence à classe de observação de treinamento mais comumente ocorrida na região a que pertence.
Na interpretação dos resultados de uma árvore de classificação, você está frequentemente interessado não apenas na previsão de classe correspondente a uma determinada região de nó terminal, mas também nas proporções de classe entre as observações de treinamento que caem nessa região.
A tarefa de crescer uma árvore de classificação é bastante semelhante à tarefa de crescer uma árvore de regressão. Assim como na regressão, você usa a divisão recursiva de binários para cultivar uma árvore de classificação. No entanto, na configuração de classificação, Soma residual dos quadrados não pode ser usada como critério para fazer as partições binárias. Em vez disso, você pode usar um destes 3 métodos abaixo:
- Classification Error Rate: Em vez de ver até que ponto uma resposta numérica está longe do valor médio, como na configuração de regressão, você pode definir a “taxa de acerto” como a fração de observações de treinamento em uma determinada região que não pertence à classe mais amplamente ocorrida. O erro é dado por esta equação:
E = 1 – argmaxc($\pi}_{mc}$)
em que $\pi}_{mc}$ representa a fração de dados de treinamento na região Rm que pertence à classe c.
- Índice de Gini: O Índice Gini é uma métrica de erro alternativa que foi concebida para mostrar o quão “pura” é uma região. “Pureza” neste caso significa quanto dos dados de treinamento em uma região em particular pertence a uma única classe. Se uma região Rm contém dados que são em sua maioria de uma única classe c então o valor do Índice Gini será pequeno:
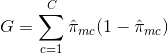
- Cross-Entropy: Uma terceira alternativa, que é semelhante ao Índice de Gini, é conhecida como Cross-Entropy ou Deviance:
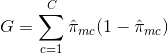
A cross-entropy assumirá um valor próximo de zero se os $\i{\i}_{mc}$’s estiverem todos próximos de 0 ou próximos de 1. Portanto, assim como o índice de Gini, a centralidade cruzada assumirá um valor pequeno se o mth nó for puro. Na verdade, acontece que o índice de Gini e a centralidade cruzada são bastante semelhantes numericamente.
Ao construir uma árvore de classificação, ou o índice de Gini ou a centralidade cruzada são tipicamente usados para avaliar a qualidade de uma determinada divisão, uma vez que são mais sensíveis à pureza do nó do que a taxa de erro de classificação. Qualquer uma destas 3 abordagens pode ser utilizada na poda da árvore, mas a taxa de erro de classificação é preferível se a precisão da previsão da árvore podada final for o objetivo.
Vantagens e desvantagens das árvores de decisão
A grande vantagem de utilizar árvores de decisão é que elas são intuitivamente muito fáceis de explicar. Elas espelham de perto a tomada de decisão humana em comparação com outras abordagens de regressão e classificação. Elas podem ser exibidas graficamente e podem facilmente lidar com preditores qualitativos sem a necessidade de criar variáveis fictícias.
No entanto, as árvores de decisão geralmente não têm o mesmo nível de precisão preditiva que outras abordagens, uma vez que não são muito robustas. Uma pequena mudança nos dados pode causar uma grande mudança na árvore estimada final.
Ao agregar muitas árvores de decisão, usando métodos como ensacamento, florestas aleatórias e aumento, o desempenho preditivo das árvores de decisão pode ser substancialmente melhorado.
Métodos baseados em árvores
Bagging
As árvores de decisão discutidas acima sofrem de alta variação, ou seja, se você dividir os dados de treinamento em 2 partes ao acaso, e ajustar uma árvore de decisão a ambas as metades, os resultados que você obtém podem ser bem diferentes. Em contraste, um procedimento com baixa variância produzirá resultados semelhantes se aplicado repetidamente a conjuntos de dados distintos.
Bagging, ou agregação bootstrap, é uma técnica usada para reduzir a variância de suas previsões através da combinação do resultado de múltiplos classificadores modelados em diferentes subamostras do mesmo conjunto de dados. Aqui está a equação para ensacar:

na qual você gera conjuntos de dados de treinamento com bootstrap diferente de $B$. Você então treina o seu método no conjunto de treinamento $bth$ bootstrapped a fim de obter $\ que (f}_{b}(x)$, e finalmente a média das previsões.
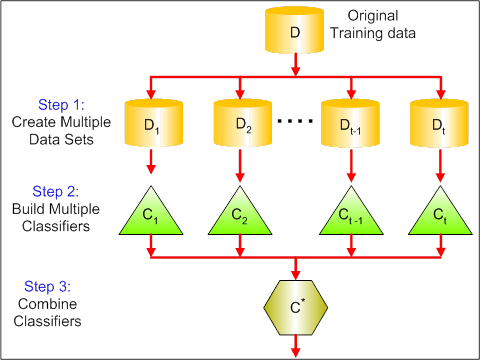
O visual abaixo mostra os 3 diferentes passos no ensacamento:
-
Passo 1: Aqui você substitui os dados originais por novos dados. Os novos dados geralmente têm uma fração das colunas e linhas dos dados originais, que então podem ser usados como hiperparâmetros no modelo de ensacamento.
-
Passo 2: Você constrói os classificadores em cada conjunto de dados. Geralmente, você pode usar o mesmo classificador para fazer modelos e previsões.
-
Passo 3: Finalmente, você usa um valor médio para combinar as previsões de todos os classificadores, dependendo do problema. Geralmente, esses valores combinados são mais robustos que um único modelo.
Embora o ensacamento possa melhorar as previsões para muitos métodos de regressão e classificação, ele é particularmente útil para árvores de decisão. Para aplicar o ensacamento em árvores de regressão/classificação, você simplesmente constrói árvores de regressão/classificação $B$ usando conjuntos de treinamento $B$ bootstrapped, e faz a média das previsões resultantes. Estas árvores são cultivadas em profundidade e não são podadas. Portanto, cada árvore individual tem alta variância, mas baixo viés. A média destas árvores $B$ reduz a variância.
Broadly falando, o ensacamento foi demonstrado para dar melhorias impressionantes na precisão, combinando centenas ou mesmo milhares de árvores em um único procedimento.
Florestas Aleatórias
Florestas Aleatórias é um método versátil de aprendizagem de máquina capaz de executar tarefas de regressão e classificação. Ele também empreende métodos de redução dimensional, trata valores faltantes, valores aberrantes e outros passos essenciais de exploração de dados, e faz um trabalho bastante bom.
Florestas Aleatórias fornece uma melhoria sobre árvores ensacadas por um pequeno ajuste que decora as árvores. Como no ensacamento, você constrói uma série de árvores de decisão em amostras de treinamento com botas. Mas ao construir estas árvores de decisão, cada vez que uma divisão em uma árvore é considerada, uma amostra aleatória de m preditores é escolhida como candidatos divididos do conjunto completo de preditores de $p$. A divisão é permitida para usar apenas um desses palpiteiros de $m$. Esta é a principal diferença entre florestas aleatórias e ensacamento; porque, como no ensacamento, a escolha do preditor $m = p$.
Para cultivar uma floresta aleatória, você deve:
-
Primeiro assumir que o número de casos no conjunto de treinamento é K. Em seguida, pegue uma amostra aleatória desses casos K e use essa amostra como o conjunto de treinamento para cultivar a árvore.
-
Se houver variáveis de input $p$, especifique um número $m < p$ de tal forma que, em cada nó, você possa selecionar variáveis aleatórias $m$ a partir do $p$. A melhor divisão desses $m$ é usada para dividir o nó.
-
Cada árvore é posteriormente cultivada na maior extensão possível e nenhuma poda é necessária.
-
Finalmente, agregue as previsões das árvores alvo para prever novos dados.
Florestas Aleatórias é muito eficaz para estimar os dados ausentes e manter a precisão quando uma grande proporção dos dados está ausente. Também pode equilibrar erros em conjuntos de dados onde as classes estão desequilibradas. Mais importante, pode lidar com grandes conjuntos de dados com grande dimensionalidade. Entretanto, uma desvantagem do uso de Florestas Aleatórias é que você pode facilmente sobreajustar conjuntos de dados ruidosos, especialmente no caso de fazer regressão.
Boosting
Boosting é outra abordagem para melhorar as previsões resultantes de uma árvore de decisão. Tal como o ensacamento e as florestas aleatórias, é uma abordagem geral que pode ser aplicada a muitos métodos de aprendizagem estatística para a regressão ou classificação. Lembre-se de que o ensacamento envolve a criação de múltiplas cópias do conjunto de dados de treinamento original usando o bootstrap, ajustando uma árvore de decisão separada a cada cópia, e depois combinando todas as árvores a fim de criar um único modelo de previsão. Notavelmente, cada árvore é construída sobre um conjunto de dados bootstrap, independente das outras árvores.
Boosting funciona de forma semelhante, exceto que as árvores são cultivadas sequencialmente: cada árvore é cultivada usando informações de árvores previamente cultivadas. Boosting não envolve amostragem bootstrap; ao invés disso, cada árvore é ajustada em uma versão modificada do conjunto de dados original.
>

Tanto para árvores de regressão como para árvores de classificação, o boosting funciona assim:
- >
Não se encaixa uma única árvore de decisão grande nos dados, o que equivale a encaixar os dados de forma dura e potencialmente sobreajustada, a abordagem de boosting aprende lentamente.
-
Dado o modelo atual, você encaixa uma árvore de decisão nos resíduos do modelo. Ou seja, você ajusta uma árvore usando os resíduos atuais, ao invés do resultado $Y$, como resposta.
-
Você então adiciona esta nova árvore de decisão na função de ajuste, a fim de atualizar os resíduos. Cada uma destas árvores pode ser bastante pequena, com apenas alguns nós terminais, determinados pelo parâmetro $d$ no algoritmo. Ajustando árvores pequenas aos resíduos, você melhora lentamente $\hat{f}$ em áreas onde não funciona bem.
-
O parâmetro de encolhimento $\nu$ torna o processo ainda mais lento, permitindo que mais e diferentes árvores de forma a atacar os resíduos.
Boosting é muito útil quando você tem muitos dados e espera que as árvores de decisão sejam muito complexas. O Boosting tem sido usado para resolver muitos problemas desafiadores de classificação e regressão, incluindo análise de risco, análise de sentimento, publicidade preditiva, modelagem de preços, estimativa de vendas e diagnóstico de pacientes, entre outros.
Árvores de decisão em R
Árvores de classificação
Para esta parte, você trabalha com o conjunto de dados Carseats usando o pacote tree em R. Tenha em mente que você precisa instalar primeiro os pacotes ISLR e tree no seu ambiente R Studio. Vamos primeiro carregar o conjunto de dados Carseats a partir do pacote ISLR.
library(ISLR)data(package="ISLR")carseats<-CarseatsVamos também carregar o pacote tree.
require(tree)O conjunto de dados Carseats é um dataframe com 400 observações sobre as 11 variáveis seguintes:
-
Vendas: vendas unitárias em milhares
-
Preço Comp: preço cobrado pelo concorrente em cada localidade
-
Income: nível de renda da comunidade em 1000s de dólares
-
Publicidade: orçamento publicitário local em cada local em 1000s de dólares
-
População: pop regional em milhares
-
Preço: preço dos assentos de carro em cada local
-
ShelveLoc: Mau, Bom ou Médio indica qualidade da localização das prateleiras
-
Idade: nível de idade da população
-
Educação: nível de ed no local
- >
Urban: Sim/Não
-
US: Sim/Não
>
>
>
names(carseats)Vamos ver o histograma de vendas de carros:
hist(carseats$Sales)Observar que Sales é uma variável quantitativa. Você quer demonstrá-la usando árvores com uma resposta binária. Para fazer isso, você transforma Sales em uma variável binária, que será chamada de High. Se a venda for inferior a 8, ela não será alta. Caso contrário, ela será alta. Então você pode colocar a nova variável High de volta no dataframe.
High = ifelse(carseats$Sales<=8, "No", "Yes")carseats = data.frame(carseats, High)Agora vamos preencher um modelo usando árvores de decisão. Claro, você não pode ter a variável Sales aqui porque a sua variável de resposta High foi criada a partir de Sales. Assim, vamos excluí-la e encaixar a árvore.
tree.carseats = tree(High~.-Sales, data=carseats)Vejamos o resumo da sua árvore de classificação:
summary(tree.carseats)Pode ver as variáveis envolvidas, o número de nós terminais, o desvio médio residual, bem como a taxa de erro de classificação. Para torná-la mais visual, vamos plotar a árvore também, depois anotá-la usando a útil text function:
plot(tree.carseats)text(tree.carseats, pretty = 0)Existem tantas variáveis, tornando muito complicado olhar para a árvore. Pelo menos, você pode ver que em cada um dos nós terminais, eles são rotulados como Yes ou No. Em cada nó de divisão, as variáveis e o valor da opção de divisão são mostrados (por exemplo, Price < 92.5 ou Advertising < 13.5).
Para um resumo detalhado da árvore, basta imprimi-la. Será útil se você quiser extrair detalhes da árvore para outros fins:
tree.carseats É hora de podar a árvore para baixo. Vamos criar um conjunto de treino e um teste dividindo o carseats dataframe em 250 treinos e 150 amostras de teste. Primeiro, você define uma semente para que os resultados sejam reprodutíveis. Em seguida, você tira uma amostra aleatória dos números de identificação (índice) das amostras. Especificamente aqui, você colhe uma amostra do conjunto 1 a n número de linhas de bancos de carro, que é 400. Você quer uma amostra de tamanho 250 (por padrão, a amostra usa sem substituição).
set.seed(101)train=sample(1:nrow(carseats), 250) Então agora você obtém este índice de train, que indexa 250 das 400 observações. Você pode reajustar o modelo com tree, usando a mesma fórmula exceto dizer à árvore para usar um subconjunto igual a train. Então vamos fazer um gráfico:
tree.carseats = tree(High~.-Sales, carseats, subset=train)plot(tree.carseats)text(tree.carseats, pretty=0)O gráfico parece um pouco diferente por causa do conjunto de dados ligeiramente diferente. No entanto, a complexidade da árvore parece aproximadamente a mesma.
Agora você vai pegar essa árvore e prever no conjunto de testes, usando o método predict para árvores. Aqui você vai querer realmente prever o class labels.
tree.pred = predict(tree.carseats, carseats, type="class")Então você pode avaliar o erro usando uma tabela de classificação errada.
with(carseats, table(tree.pred, High))Nas diagonais estão as classificações corretas, enquanto fora das diagonais estão as incorretas. Você só quer registrar as corretas. Para fazer isso, você pode pegar a soma das 2 diagonais divididas pelo total (150 observações de teste).
(72 + 43) / 150Ok, você obtém um erro de 0,76 com esta árvore.
Ao crescer uma grande árvore arbustiva, ela pode ter muita variação. Assim, vamos usar a validação cruzada para podar a árvore de forma otimizada. Usando cv.tree, você usará o erro de classificação errada como base para fazer a poda.
cv.carseats = cv.tree(tree.carseats, FUN = prune.misclass)cv.carseatsImprimir os resultados mostra os detalhes do caminho da validação cruzada. Você pode ver os tamanhos das árvores à medida que foram podadas, os desvios à medida que a poda prosseguiu, bem como o parâmetro de complexidade de custo utilizado no processo.
Vejamos o plot out:
plot(cv.carseats)Vejamos a parte em espiral descendente devido ao erro de classificação errada em 250 pontos de validação cruzada. Então vamos escolher um valor nos passos para baixo (12). Então, vamos podar a árvore até um tamanho de 12 para identificar essa árvore. Finalmente, vamos plotar e anotar essa árvore para ver o resultado.
prune.carseats = prune.misclass(tree.carseats, best = 12)plot(prune.carseats)text(prune.carseats, pretty=0)É um pouco mais raso que as árvores anteriores, e você pode realmente ler as etiquetas. Vamos avaliar novamente no conjunto de dados do teste.
tree.pred = predict(prune.carseats, carseats, type="class")with(carseats, table(tree.pred, High))(74 + 39) / 150Parece que as classificações corretas caíram um pouco. Fez mais ou menos o mesmo que a sua árvore original, então a poda não doeu muito no que diz respeito a erros de classificação errados, e deu uma árvore mais simples.
A maior parte das vezes, as árvores não dão erros de previsão muito bons, então vamos em frente dar uma olhada nas florestas aleatórias e no boosting, que tendem a superar as árvores no que diz respeito à previsão e ao erro de classificação.
Florestas aleatórias
Para esta parte, você vai usar o Boston housing data para explorar as florestas aleatórias e o boosting. O conjunto de dados está localizado no pacote MASS. Ele dá valores de habitação e outras estatísticas em cada um dos 506 subúrbios de Boston com base em um censo de 1970.
library(MASS)data(package="MASS")boston<-Bostondim(boston)names(boston)Let’s também carregam o pacote randomForest.
require(randomForest)Para preparar dados para floresta aleatória, vamos definir a semente e criar uma amostra de treinamento de 300 observações.
set.seed(101)train = sample(1:nrow(boston), 300) Neste conjunto de dados, há 506 surburbs de Boston. Para cada surburb, você tem variáveis como crime per capita, tipos de indústria, média # de quartos por habitação, proporção média de idade das casas, etc. Vamos usar medv – o valor mediano das casas ocupadas pelos proprietários para cada um desses surburbos, como a variável de resposta.
Vejamos como ela se encaixa em uma floresta aleatória e vejamos como ela funciona bem. Como foi dito, você usa a resposta medv, o valor mediano da habitação (em dólares de $1K), e o conjunto de amostras de treinamento.
rf.boston = randomForest(medv~., data = boston, subset = train)rf.bostonPrinting out the random forest gives its summary: the # of trees (500 were grown), the mean squared residuals (MSR), and the percentage of variance explained. O MSR e a % de variância explicada são baseados nas estimativas fora do saco, um dispositivo muito inteligente em florestas aleatórias para obter estimativas de erro honestas.
O único parâmetro de ajuste em uma floresta aleatória é o argumento chamado mtry, que é o número de variáveis que são selecionadas em cada divisão de cada árvore quando você faz uma divisão. Como visto aqui, mtry é 4 das 13 variáveis exploratórias (excluindo medv) nos dados de Boston Housing – o que significa que cada vez que a árvore vem para dividir um nó, 4 variáveis seriam selecionadas aleatoriamente, então a divisão estaria confinada a 1 dessas 4 variáveis. É assim que randomForests descorrelata as árvores.
Vai caber uma série de florestas aleatórias. Existem 13 variáveis, então vamos ter mtry intervalo de 1 a 13:
-
Para registrar os erros, você configura 2 variáveis
oob.erretest.err. -
Num loop de
mtryde 1 a 13, você primeiro encaixa orandomForestcom aquele valor demtryno conjunto de dadostrain, restringindo o número de árvores a ser 350. -
Então você extrai o erro médio-quadrado no objeto (o erro fora do saco).
-
Então você prevê no conjunto de dados do teste (
boston) usandofit(o ajuste derandomForest). -
Por último, você calcula o erro do teste: erro médio-quadrado, que é igual a
mean( (medv - pred) ^ 2 ).
oob.err = double(13)test.err = double(13)for(mtry in 1:13){ fit = randomForest(medv~., data = boston, subset=train, mtry=mtry, ntree = 350) oob.err = fit$mse pred = predict(fit, boston) test.err = with(boston, mean( (medv-pred)^2 ))}Basicamente você acabou de cultivar 4550 árvores (13 vezes 350). Agora vamos fazer um gráfico usando o comando matplot. O erro de teste e o erro fora do saco são unidos para fazer uma matriz de 2 colunas. Existem alguns outros argumentos na matriz, incluindo os valores dos caracteres de plotagem (pch = 23 significa diamante preenchido), cores (vermelho e azul), tipo igual a ambos (plotagem de ambos os pontos e conexão com as linhas), e nome do eixo y (erro médio quadrático). Você também pode colocar uma legenda no canto superior direito do gráfico.
matplot(1:mtry, cbind(test.err, oob.err), pch = 23, col = c("red", "blue"), type = "b", ylab="Mean Squared Error")legend("topright", legend = c("OOB", "Test"), pch = 23, col = c("red", "blue"))Idealmente, estas 2 curvas devem alinhar-se, mas parece que o erro de teste é um pouco mais baixo. No entanto, há uma grande variabilidade nestas estimativas de erros de teste. Como a estimativa de erro fora do saco foi calculada em um conjunto de dados e a estimativa de erro de teste foi calculada em outro conjunto de dados, estas diferenças estão bem dentro dos erros padrão.
Notem que a curva vermelha está suavemente acima da curva azul? Estas estimativas de erros estão muito correlacionadas, porque a randomForest com mtry = 4 é muito parecida com a que tem mtry = 5. É por isso que cada uma das curvas é bastante lisa. O que você vê é que mtry com mtry = 4 parece ser a escolha mais ótima, pelo menos para o erro de teste. Este valor de mtry para o erro fora do saco é igual a 9,
Então com muito poucas camadas, você ajustou um modelo de previsão muito poderoso usando florestas aleatórias. Como assim? O lado esquerdo mostra o desempenho de uma única árvore. O erro médio ao quadrado no out-of-bag é 26, e você caiu para cerca de 15 (apenas um pouco acima da metade). Isto significa que você reduziu o erro pela metade. Da mesma forma para o erro de teste, você reduziu o erro de 20 para 12,
Boosting
Comparado a florestas aleatórias, aumentando o crescimento de árvores menores e mais toco e vai no viés. Você usará o pacote GBM (Gradient Boosted Modeling), em R.
require(gbm)GBM pede a distribuição, que é gaussiana, porque você estará fazendo a perda de erro ao quadrado. Você vai pedir GBM por 10.000 árvores, o que soa como muito, mas estas serão árvores rasas. A profundidade de interação é o número de partições, então você quer 4 partições em cada árvore. O encolhimento é de 0,01, que é o quanto você vai encolher a árvore passo atrás.
boost.boston = gbm(medv~., data = boston, distribution = "gaussian", n.trees = 10000, shrinkage = 0.01, interaction.depth = 4)summary(boost.boston)A função summary dá um gráfico de importância variável. Parece que existem 2 variáveis que têm uma grande importância relativa: rm (número de divisões) e lstat (percentagem de pessoas de menor estatuto económico na comunidade). Vamos traçar estas 2 variáveis:
plot(boost.boston,i="lstat")plot(boost.boston,i="rm")O primeiro gráfico mostra que quanto maior a proporção de pessoas de menor estatuto económico no subúrbio, menor o valor dos preços das habitações. O 2º gráfico mostra a relação inversa com o número de quartos: o número médio de quartos na casa aumenta à medida que o preço aumenta.
É hora de prever um modelo reforçado no conjunto de dados do teste. Vamos ver o desempenho do teste como uma função do número de árvores:
-
Primeiro, você faz uma grade de número de árvores em passos de 100 de 100 para 10.000.
-
Então, você executa a função
predictno modelo impulsionado. Ela toman.treescomo argumento, e produz uma matriz de predições nos dados do teste. -
As dimensões da matriz são 206 observações de teste e 100 diferentes vetores de predição nos 100 diferentes valores da árvore.
n.trees = seq(from = 100, to = 10000, by = 100)predmat = predict(boost.boston, newdata = boston, n.trees = n.trees)dim(predmat)
É hora de calcular o erro do teste para cada um dos vetores preditores:
-
predmaté uma matriz,medvé um vetor, portanto (predmat–medv) é uma matriz de diferenças. Você pode usar a funçãoapplypara as colunas dessas diferenças quadradas (a média). Isso computaria o erro quadrático médio da coluna para os vetores de previsão. -
Então você faz um gráfico usando parâmetros similares ao usado para a Floresta Aleatória. Ele mostraria um gráfico de erro de boosting.
boost.err = with(boston, apply( (predmat - medv)^2, 2, mean) )plot(n.trees, boost.err, pch = 23, ylab = "Mean Squared Error", xlab = "# Trees", main = "Boosting Test Error")abline(h = min(test.err), col = "red")
O erro de boosting diminui à medida que o número de árvores aumenta. Esta é uma evidência que mostra que o boosting é relutante em se ajustar em excesso. Vamos também incluir o melhor erro de teste da randomForest na trama. O boosting na verdade recebe uma quantidade razoável abaixo do erro de teste do randomForest.
Conclusion
Então este é o fim deste tutorial R sobre a construção de modelos de árvores de decisão: árvores de classificação, florestas aleatórias, e árvores de boost. Os últimos 2 são métodos poderosos que você pode usar a qualquer momento, conforme necessário. Na minha experiência, o RandomForest tem um melhor desempenho do que o RandomForest, mas o RandomForest é mais fácil de implementar. No RandomForest, o único parâmetro de afinação é o número de árvores; enquanto no reforço, mais parâmetros de afinação são necessários além do número de árvores, incluindo a retração e a profundidade de interação.
Se você gostaria de aprender mais, não deixe de dar uma olhada no nosso curso Caixa de Ferramentas de Aprendizagem de Máquinas para R.