Introduction
L’arbre de décision est un type d’algorithme d’apprentissage supervisé qui peut être utilisé dans les problèmes de régression et de classification. Il fonctionne pour les variables d’entrée et de sortie à la fois catégoriques et continues.
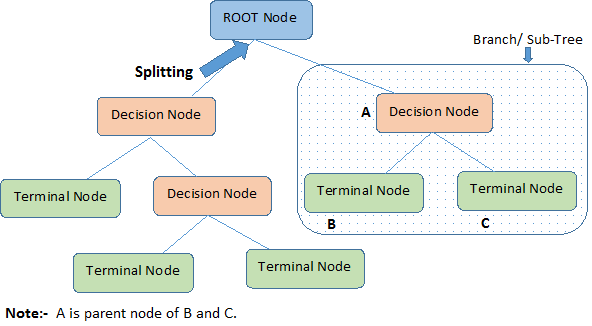
Identifions les terminologies importantes sur l’arbre de décision, en regardant l’image ci-dessus :
-
Le nœud racine représente la population ou l’échantillon entier. Il se divise ensuite en deux ou plusieurs ensembles homogènes.
-
Le fractionnement est un processus de division d’un nœud en deux ou plusieurs sous-nœuds.
-
Lorsqu’un sous-nœud se divise en d’autres sous-nœuds, il est appelé nœud de décision.
-
Les nœuds qui ne se divisent pas sont appelés nœud terminal ou feuille.
-
Lorsque vous supprimez les sous-nœuds d’un nœud de décision, ce processus est appelé élagage. Le contraire de l’élagage est le fractionnement.
-
Une sous-section d’un arbre entier est appelée branche.
-
Un nœud, qui est divisé en sous-nœuds est appelé nœud parent des sous-nœuds ; tandis que les sous-nœuds sont appelés enfant du nœud parent.
Types d’arbres de décision
Arbres de régression
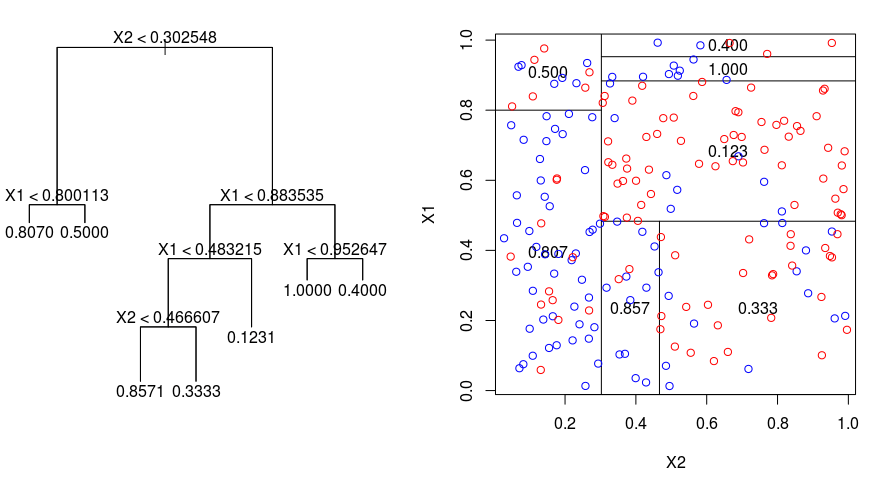
Regardons l’image ci-dessous, qui aide à visualiser la nature du partitionnement effectué par un arbre de régression. Elle montre un arbre non élagué et un arbre de régression ajusté à un ensemble de données aléatoires. Les deux visualisations montrent une série de règles de partitionnement, en commençant par le sommet de l’arbre. Remarquez que chaque division du domaine est alignée avec l’un des axes des caractéristiques. Le concept de division parallèle à l’axe se généralise directement aux dimensions supérieures à deux. Pour un espace de caractéristiques de taille $p$, un sous-ensemble de $\mathbb{R}^p$, l’espace est divisé en $M$ régions, $R_{m}$, dont chacune est un « hyperbloc » de dimension $p$.
Pour construire un arbre de régression, vous utilisez d’abord la division binaire récursive pour faire croître un grand arbre sur les données d’apprentissage, en ne s’arrêtant que lorsque chaque nœud terminal a moins d’un certain nombre minimum d’observations. Le fractionnement binaire récursif est un algorithme gourmand et descendant utilisé pour minimiser la somme résiduelle des carrés (RSS), une mesure d’erreur également utilisée dans les paramètres de régression linéaire. Le RSS, dans le cas d’un espace caractéristique partitionné avec M partitions est donné par :
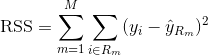
En commençant au sommet de l’arbre, vous le divisez en 2 branches, créant une partition de 2 espaces. Vous effectuez ensuite cette division particulière au sommet de l’arbre plusieurs fois et choisissez la division des caractéristiques qui minimise le RSS (actuel).
Puis, vous appliquez l’élagage de la complexité des coûts au grand arbre afin d’obtenir une séquence des meilleurs sous-arbres, en fonction de $\alpha$. L’idée de base ici est d’introduire un paramètre de réglage supplémentaire, noté $\alpha$, qui équilibre la profondeur de l’arbre et sa qualité d’ajustement aux données d’apprentissage.
Vous pouvez utiliser la validation croisée K-fold pour choisir $\alpha$. Cette technique consiste simplement à diviser les observations d’apprentissage en K plis pour estimer le taux d’erreur de test des sous-arbres. Votre objectif est de sélectionner celui qui conduit au taux d’erreur le plus faible.
Arbres de classification
Un arbre de classification est très similaire à un arbre de régression, sauf qu’il est utilisé pour prédire une réponse qualitative plutôt que quantitative.
Rappellez-vous que pour un arbre de régression, la réponse prédite pour une observation est donnée par la réponse moyenne des observations d’entraînement qui appartiennent au même nœud terminal. En revanche, pour un arbre de classification, vous prédisez que chaque observation appartient à la classe d’observations d’entraînement la plus fréquente dans la région à laquelle elle appartient.
En interprétant les résultats d’un arbre de classification, vous êtes souvent intéressé non seulement par la prédiction de classe correspondant à une région de nœud terminal particulière, mais aussi par les proportions de classe parmi les observations d’entraînement qui tombent dans cette région.
La tâche de croissance d’un arbre de classification est assez similaire à la tâche de croissance d’un arbre de régression. Tout comme dans le cadre de la régression, vous utilisez le fractionnement binaire récursif pour faire croître un arbre de classification. Cependant, dans le cadre de la classification, la somme résiduelle des carrés ne peut pas être utilisée comme critère pour effectuer les divisions binaires. Au lieu de cela, vous pouvez utiliser l’une de ces 3 méthodes ci-dessous :
- Taux d’erreur de classification : Plutôt que de voir à quel point une réponse numérique est éloignée de la valeur moyenne, comme dans le paramètre de régression, vous pouvez plutôt définir le « taux d’erreur » comme la fraction des observations de formation dans une région particulière qui n’appartiennent pas à la classe la plus répandue. L’erreur est donnée par cette équation :
E = 1 – argmaxc($\hat{\pi}_{mc}$)
dans laquelle $\hat{\pi}_{mc}$ représente la fraction des données d’apprentissage dans la région Rm qui appartiennent à la classe c.
- Indice de Gini : L’indice de Gini est une métrique d’erreur alternative qui est conçue pour montrer à quel point une région est « pure ». « Pureté » dans ce cas signifie combien de données d’entraînement dans une région particulière appartiennent à une seule classe. Si une région Rm contient des données provenant principalement d’une seule classe c, la valeur de l’indice de Gini sera faible :
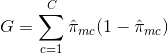
- L’entropie croisée : Une troisième alternative, qui est similaire à l’indice de Gini, est connue sous le nom d’entropie croisée ou déviance :
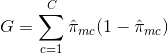
L’entropie croisée prendra une valeur proche de zéro si les $\hat{\pi}_{mc}$ sont tous proches de 0 ou proches de 1. Par conséquent, comme l’indice de Gini, l’entropie croisée prendra une petite valeur si le mième nœud est pur. En fait, il s’avère que l’indice de Gini et l’entropie croisée sont assez similaires numériquement.
Lors de la construction d’un arbre de classification, l’indice de Gini ou l’entropie croisée sont généralement utilisés pour évaluer la qualité d’une division particulière, car ils sont plus sensibles à la pureté des nœuds que le taux d’erreur de classification. Chacune de ces 3 approches pourrait être utilisée lors de l’élagage de l’arbre, mais le taux d’erreur de classification est préférable si la précision de prédiction de l’arbre final élagué est l’objectif.
Avantages et inconvénients des arbres de décision
Le principal avantage de l’utilisation des arbres de décision est qu’ils sont intuitivement très faciles à expliquer. Ils reflètent étroitement la prise de décision humaine par rapport aux autres approches de régression et de classification. Ils peuvent être affichés graphiquement, et ils peuvent facilement gérer les prédicteurs qualitatifs sans avoir besoin de créer des variables fictives.
Cependant, les arbres de décision n’ont généralement pas le même niveau de précision prédictive que les autres approches, car ils ne sont pas tout à fait robustes. Un petit changement dans les données peut entraîner un grand changement dans l’arbre final estimé.
En agrégeant de nombreux arbres de décision, en utilisant des méthodes comme le bagging, les forêts aléatoires et le boosting, la performance prédictive des arbres de décision peut être considérablement améliorée.
Méthodes basées sur les arbres
Bagging
Les arbres de décision discutés ci-dessus souffrent d’une variance élevée, ce qui signifie que si vous divisez les données d’entraînement en 2 parties au hasard, et que vous ajustez un arbre de décision aux deux moitiés, les résultats que vous obtenez pourraient être très différents. En revanche, une procédure à faible variance donnera des résultats similaires si elle est appliquée de manière répétée à un ensemble de données distinct.
Le bagging, ou agrégation bootstrap, est une technique utilisée pour réduire la variance de vos prédictions en combinant le résultat de plusieurs classificateurs modélisés sur différents sous-échantillons du même ensemble de données. Voici l’équation du bagging :

dans lequel vous générez $B$ différents ensembles de données d’entraînement bootstrap. Vous entraînez ensuite votre méthode sur le $bth$ ensemble d’entraînement bootstrapped afin d’obtenir $\hat{f}_{b}(x)$, et enfin vous faites la moyenne des prédictions.
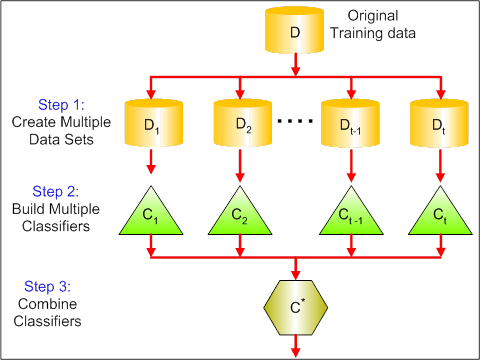
Le visuel ci-dessous montre les 3 différentes étapes du bagging:
-
Etape 1 : Ici, vous remplacez les données originales par de nouvelles données. Les nouvelles données ont généralement une fraction des colonnes et des lignes des données originales, qui peuvent ensuite être utilisées comme hyper-paramètres dans le modèle de bagging.
-
Étape 2 : Vous construisez des classificateurs sur chaque ensemble de données. Généralement, vous pouvez utiliser le même classificateur pour faire des modèles et des prédictions.
-
Étape 3 : Enfin, vous utilisez une valeur moyenne pour combiner les prédictions de tous les classificateurs, selon le problème. En général, ces valeurs combinées sont plus robustes qu’un modèle unique.
Bien que le bagging puisse améliorer les prédictions de nombreuses méthodes de régression et de classification, il est particulièrement utile pour les arbres de décision. Pour appliquer le bagging aux arbres de régression/classification, il suffit de construire $B$ arbres de régression/classification en utilisant $B$ ensembles d’entraînement bootstrapped, et de faire la moyenne des prédictions résultantes. Ces arbres sont développés en profondeur et ne sont pas élagués. Par conséquent, chaque arbre individuel a une variance élevée, mais un faible biais. Le fait de faire la moyenne de ces $B$ arbres réduit la variance.
En gros, il a été démontré que le bagging donne des améliorations impressionnantes de la précision en combinant ensemble des centaines, voire des milliers d’arbres dans une seule procédure.
Forêts aléatoires
Les forêts aléatoires sont une méthode d’apprentissage automatique polyvalente capable d’effectuer des tâches de régression et de classification. Elle entreprend également des méthodes de réduction dimensionnelle, traite les valeurs manquantes, les valeurs aberrantes et d’autres étapes essentielles de l’exploration des données, et fait un assez bon travail.
Les Forêts aléatoires apportent une amélioration par rapport aux arbres en sac par une petite astuce qui décorrèle les arbres. Comme dans la mise en sac, vous construisez un certain nombre d’arbres de décision sur des échantillons d’entraînement bootstrapped. Mais lors de la construction de ces arbres de décision, chaque fois qu’une division dans un arbre est envisagée, un échantillon aléatoire de m prédicteurs est choisi comme candidats à la division parmi l’ensemble complet de $p$ prédicteurs. La division ne peut utiliser qu’un seul de ces $m$ prédicteurs. C’est la principale différence entre les forêts aléatoires et le bagging ; car comme dans le bagging, le choix du prédicteur $m = p$.
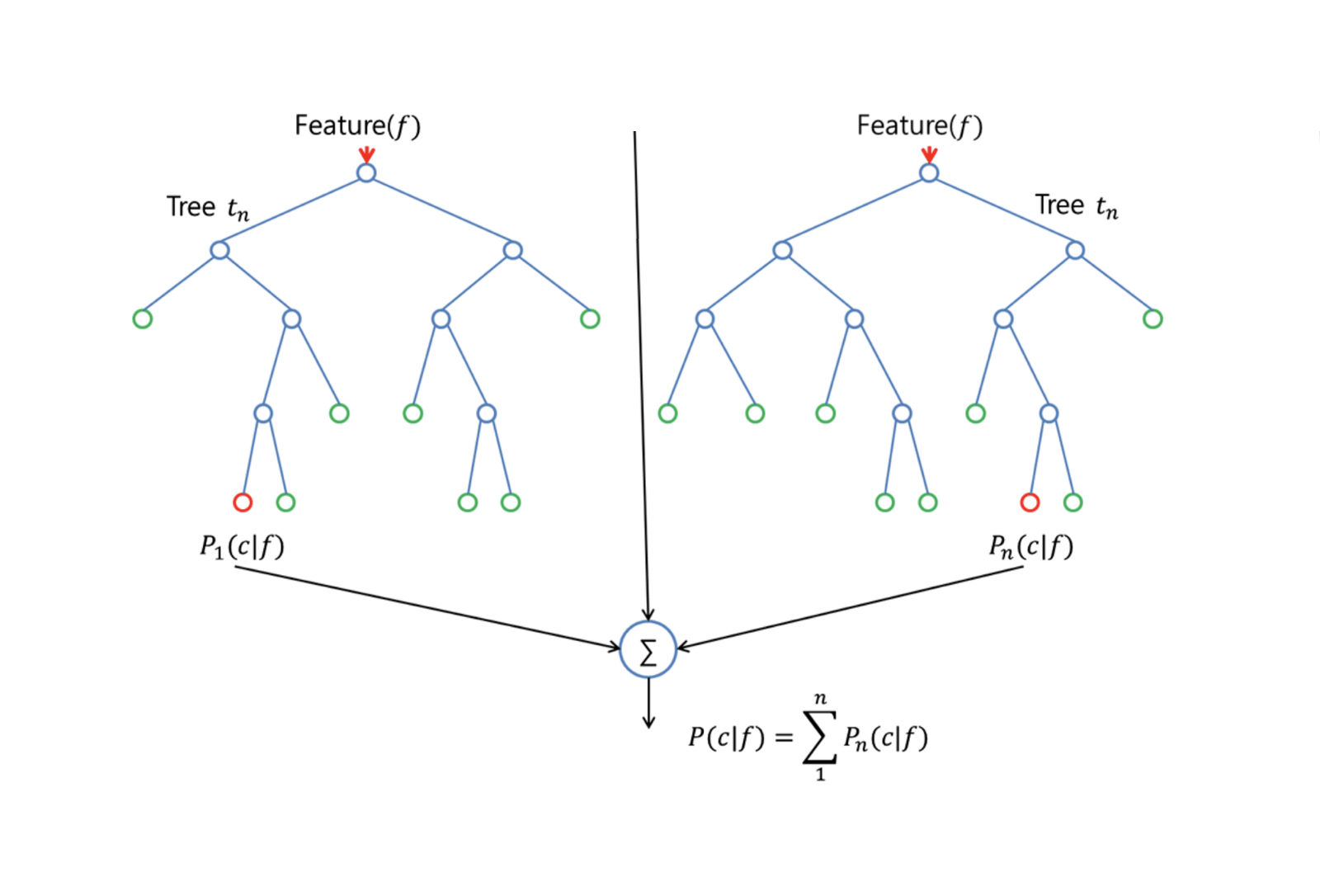
Pour faire croître une forêt aléatoire, vous devez :
-
Premièrement, supposer que le nombre de cas dans l’ensemble d’entraînement est K. Ensuite, prenez un échantillon aléatoire de ces K cas, puis utilisez cet échantillon comme ensemble d’entraînement pour faire croître l’arbre.
-
S’il y a $p$ variables d’entrée, spécifiez un nombre $m < p$ tel qu’à chaque nœud, vous pouvez sélectionner $m$ variables aléatoires parmi les $p$. La meilleure division sur ces $m$ est utilisée pour diviser le nœud.
-
Chaque arbre est par la suite développé au maximum et aucun élagage n’est nécessaire.
-
Enfin, agréger les prédictions des arbres cibles pour prédire de nouvelles données.
Les forêts aléatoires sont très efficaces pour estimer les données manquantes et maintenir la précision lorsqu’une grande partie des données est manquante. Il peut également équilibrer les erreurs dans les ensembles de données où les classes sont déséquilibrées. Plus important encore, il peut traiter des ensembles de données massifs avec une grande dimensionnalité. Cependant, l’un des inconvénients de l’utilisation des forêts aléatoires est que vous pourriez facilement surajuster des ensembles de données bruyants, en particulier dans le cas de faire de la régression.
Boosting
Le boosting est une autre approche pour améliorer les prédictions résultant d’un arbre de décision. Comme le bagging et les forêts aléatoires, il s’agit d’une approche générale qui peut être appliquée à de nombreuses méthodes d’apprentissage statistique pour la régression ou la classification. Rappelons que la mise en sac implique la création de plusieurs copies de l’ensemble de données de formation original à l’aide du bootstrap, l’adaptation d’un arbre de décision distinct à chaque copie, puis la combinaison de tous les arbres afin de créer un modèle prédictif unique. Notamment, chaque arbre est construit sur un ensemble de données bootstrap, indépendamment des autres arbres.
Le boosting fonctionne de manière similaire, sauf que les arbres sont cultivés séquentiellement : chaque arbre est cultivé en utilisant les informations des arbres cultivés précédemment. Le boosting n’implique pas d’échantillonnage bootstrap ; au lieu de cela, chaque arbre est ajusté sur une version modifiée de l’ensemble de données original.

Pour les arbres de régression et de classification, le boosting fonctionne comme suit :
-
Contrairement à l’ajustement d’un seul grand arbre de décision aux données, qui revient à ajuster les données de manière stricte et potentiellement à les surajuster, l’approche de boosting apprend plutôt lentement.
-
Selon le modèle actuel, vous ajustez un arbre de décision aux résidus du modèle. C’est-à-dire que vous ajustez un arbre en utilisant les résidus actuels, plutôt que le résultat $Y$, comme réponse.
-
Vous ajoutez ensuite ce nouvel arbre de décision dans la fonction ajustée afin de mettre à jour les résidus. Chacun de ces arbres peut être assez petit, avec seulement quelques nœuds terminaux, déterminés par le paramètre $d$ dans l’algorithme. En ajustant de petits arbres aux résidus, vous améliorez lentement $\hat{f}$ dans les domaines où il n’est pas performant.
-
Le paramètre de rétrécissement $\nu$ ralentit encore plus le processus, permettant à des arbres plus nombreux et de formes différentes d’attaquer les résidus.
Le boosting est très utile lorsque vous avez beaucoup de données et que vous vous attendez à ce que les arbres de décision soient très complexes. Le boosting a été utilisé pour résoudre de nombreux problèmes difficiles de classification et de régression, y compris l’analyse des risques, l’analyse des sentiments, la publicité prédictive, la modélisation des prix, l’estimation des ventes et le diagnostic des patients, entre autres.
Arbres de décision dans R
Arbres de classification
Pour cette partie, vous travaillez avec le jeu de données Carseats en utilisant le paquet tree dans R. Gardez à l’esprit que vous devez d’abord installer les paquets ISLR et tree dans votre environnement R Studio. Chargeons d’abord le cadre de données Carseats à partir du paquet ISLR.
library(ISLR)data(package="ISLR")carseats<-CarseatsChargeons également le paquet tree.
require(tree)Le jeu de données Carseats est un dataframe avec 400 observations sur les 11 variables suivantes:
-
Ventes : ventes unitaires en milliers
-
CompPrice : prix pratiqué par le concurrent à chaque emplacement
-
Revenu : niveau de revenu de la communauté en milliers de dollars
-
Publicité : budget publicitaire local à chaque emplacement en 1000s de dollars
-
Population : pop régionale en milliers
-
Prix : prix des sièges auto à chaque emplacement
-
ShelveLoc : Mauvais, Bon ou Moyen indique la qualité de l’emplacement des étagères
-
Age : niveau d’âge de la population
-
Éducation : niveau d’ed à l’emplacement
-
Urbain : Oui/Non
-
US : Oui/Non
names(carseats)Regardons l’histogramme des ventes de voitures:
hist(carseats$Sales)Observez que Sales est une variable quantitative. Vous voulez la démontrer en utilisant des arbres avec une réponse binaire. Pour ce faire, vous transformez Sales en une variable binaire, qui sera appelée High. Si le chiffre d’affaires est inférieur à 8, il ne sera pas élevé. Dans le cas contraire, il sera élevé. Ensuite, vous pouvez remettre cette nouvelle variable High dans le dataframe.
High = ifelse(carseats$Sales<=8, "No", "Yes")carseats = data.frame(carseats, High)Maintenant, remplissons un modèle en utilisant des arbres de décision. Bien sûr, vous ne pouvez pas avoir la variable Sales ici parce que votre variable de réponse High a été créée à partir de Sales. Ainsi, excluons-la et ajustons l’arbre.
tree.carseats = tree(High~.-Sales, data=carseats)Voyons le résumé de votre arbre de classification :
summary(tree.carseats)Vous pouvez voir les variables impliquées, le nombre de nœuds terminaux, la déviance moyenne résiduelle, ainsi que le taux d’erreur de mauvaise classification. Pour rendre cela plus visuel, traçons également l’arbre, puis annotons-le à l’aide de la très pratique fonction text:
plot(tree.carseats)text(tree.carseats, pretty = 0)Il y a tellement de variables, qu’il est très compliqué de regarder l’arbre. Au moins, vous pouvez voir qu’à chacun des nœuds terminaux, elles sont étiquetées Yes ou No. À chaque nœud de fractionnement, les variables et la valeur du choix de fractionnement sont indiquées (par exemple, Price < 92.5 ou Advertising < 13.5).
Pour un résumé détaillé de l’arbre, il suffit de l’imprimer. Il sera pratique si vous voulez extraire des détails de l’arbre à d’autres fins :
tree.carseatsIl est temps d’élaguer l’arbre. Créons un ensemble de formation et un test en divisant la trame de données carseats en 250 échantillons de formation et 150 échantillons de test. Tout d’abord, vous définissez une graine pour que les résultats soient reproductibles. Ensuite, vous prenez un échantillon aléatoire des numéros d’identification (index) des échantillons. Dans ce cas précis, vous prélevez un échantillon dans l’ensemble de 1 à n rangées de sièges de voiture, soit 400. Vous voulez un échantillon de taille 250 (par défaut, l’échantillon utilise sans remplacement).
set.seed(101)train=sample(1:nrow(carseats), 250)Donc maintenant vous obtenez cet indice de train, qui indexe 250 des 400 observations. Vous pouvez refaire le modèle avec tree, en utilisant la même formule sauf en disant à l’arbre d’utiliser un sous-ensemble égale train. Ensuite, faisons un tracé :
tree.carseats = tree(High~.-Sales, carseats, subset=train)plot(tree.carseats)text(tree.carseats, pretty=0)Le tracé semble un peu différent en raison de l’ensemble de données légèrement différent. Néanmoins, la complexité de l’arbre semble à peu près la même.
Maintenant, vous allez prendre cet arbre et le prédire sur l’ensemble de test, en utilisant la méthode predict pour les arbres. Ici, vous voudrez réellement prédire les étiquettes class.
tree.pred = predict(tree.carseats, carseats, type="class")Puis vous pouvez évaluer l’erreur en utilisant une table de mauvaise classification.
with(carseats, table(tree.pred, High))Sur les diagonales sont les classifications correctes, tandis que hors des diagonales sont les incorrectes. Vous voulez seulement recorriger les correctes. Pour cela, vous pouvez prendre la somme des 2 diagonales divisée par le total (150 observations de test).
(72 + 43) / 150Ok, vous obtenez une erreur de 0,76 avec cet arbre.
Lorsqu’on fait pousser un grand arbre touffu, il pourrait avoir trop de variance. Ainsi, utilisons la validation croisée pour élaguer l’arbre de manière optimale. En utilisant cv.tree, vous utiliserez l’erreur de mauvaise classification comme base pour faire l’élagage.
cv.carseats = cv.tree(tree.carseats, FUN = prune.misclass)cv.carseatsL’impression des résultats montre les détails du chemin de la validation croisée. Vous pouvez voir les tailles des arbres au fur et à mesure de leur élagage, les écarts au fur et à mesure de l’élagage, ainsi que le paramètre de complexité des coûts utilisé dans le processus.
Traçons cela :
plot(cv.carseats)En regardant le tracé, vous voyez une partie en spirale descendante à cause de l’erreur de classification sur 250 points de validation croisée. Choisissons donc une valeur dans les marches descendantes (12). Ensuite, nous élaguons l’arbre à une taille de 12 pour identifier cet arbre. Enfin, traçons et annotons cet arbre pour voir le résultat.
prune.carseats = prune.misclass(tree.carseats, best = 12)plot(prune.carseats)text(prune.carseats, pretty=0)Il est un peu moins profond que les arbres précédents, et vous pouvez réellement lire les étiquettes. Évaluons-le à nouveau sur le jeu de données de test.
tree.pred = predict(prune.carseats, carseats, type="class")with(carseats, table(tree.pred, High))(74 + 39) / 150Il semble que les classifications correctes aient un peu diminué. Il a fait à peu près la même chose que votre arbre original, donc l’élagage n’a pas fait beaucoup de mal en ce qui concerne les erreurs de classification, et a donné un arbre plus simple.
Souvent, les arbres ne donnent pas de très bonnes erreurs de prédiction, alors allons-y, jetons un coup d’œil aux forêts aléatoires et au boosting, qui ont tendance à surpasser les arbres en ce qui concerne la prédiction et les erreurs de classification.
Forêts aléatoires
Pour cette partie, vous utiliserez le Boston housing data pour explorer les forêts aléatoires et le boosting. Le jeu de données est situé dans le paquetage MASS. Il donne les valeurs des logements et d’autres statistiques dans chacune des 506 banlieues de Boston sur la base d’un recensement de 1970.
library(MASS)data(package="MASS")boston<-Bostondim(boston)names(boston)Chargeons également le paquet randomForest.
require(randomForest)Pour préparer les données pour la forêt aléatoire, définissons la graine et créons un ensemble d’entraînement échantillon de 300 observations.
set.seed(101)train = sample(1:nrow(boston), 300)Dans ce jeu de données, il y a 506 banlieues de Boston. Pour chaque banlieue, vous avez des variables telles que le crime par habitant, les types d’industrie, le # moyen de pièces par logement, la proportion moyenne de l’âge des maisons, etc. Utilisons medv – la valeur médiane des maisons occupées par leur propriétaire pour chacun de ces surburbs, comme variable de réponse.
Envisageons une forêt aléatoire et voyons comment elle se comporte. Comme cela a été dit, vous utilisez la réponse medv, la valeur médiane des logements (en dollars $1K), et l’ensemble d’échantillons d’entraînement.
rf.boston = randomForest(medv~., data = boston, subset = train)rf.bostonL’impression de la forêt aléatoire donne son résumé : le # d’arbres (500 ont été cultivés), les résidus quadratiques moyens (MSR), et le pourcentage de variance expliquée. Le MSR et le % de variance expliquée sont basés sur les estimations out-of-bag, un dispositif très astucieux dans les forêts aléatoires pour obtenir des estimations d’erreur honnêtes.
Le seul paramètre de réglage dans une forêt aléatoire est l’argument appelé mtry, qui est le nombre de variables qui sont sélectionnées à chaque fractionnement de chaque arbre lorsque vous faites un fractionnement. Comme on le voit ici, mtry est 4 des 13 variables exploratoires (à l’exclusion de medv) dans les données de Boston Housing – ce qui signifie que chaque fois que l’arbre vient à fractionner un nœud, 4 variables seraient sélectionnées au hasard, puis le fractionnement serait limité à 1 de ces 4 variables. C’est ainsi que randomForests dé-corrèle les arbres.
Vous allez ajuster une série de forêts aléatoires. Il y a 13 variables, donc faisons en sorte que mtry varie de 1 à 13:
-
Pour enregistrer les erreurs, vous mettez en place 2 variables
oob.errettest.err. -
Dans une boucle de
mtryde 1 à 13, vous ajustez d’abord lerandomForestavec cette valeur demtrysur le jeu de donnéestrain, en limitant le nombre d’arbres à 350. -
Puis vous extrayez l’erreur quadratique moyenne sur l’objet (l’erreur hors du sac).
-
Puis vous prédisez sur le jeu de données de test (
boston) en utilisantfit(l’ajustement derandomForest). -
En dernier lieu, vous calculez l’erreur de test : erreur quadratique moyenne, qui est égale à
mean( (medv - pred) ^ 2 ).
oob.err = double(13)test.err = double(13)for(mtry in 1:13){ fit = randomForest(medv~., data = boston, subset=train, mtry=mtry, ntree = 350) oob.err = fit$mse pred = predict(fit, boston) test.err = with(boston, mean( (medv-pred)^2 ))}En gros, vous venez de faire pousser 4550 arbres (13 fois 350). Maintenant, faisons un tracé en utilisant la commande matplot. L’erreur de test et l’erreur de sortie de sac sont liées ensemble pour faire une matrice à 2 colonnes. Il y a quelques autres arguments dans la matrice, y compris les valeurs des caractères de traçage (pch = 23 signifie un diamant rempli), les couleurs (rouge et bleu), le type est égal à both (tracer les deux points et les relier avec les lignes), et le nom de l’axe des y (Mean Squared Error). Vous pouvez également mettre une légende dans le coin supérieur droit du tracé.
matplot(1:mtry, cbind(test.err, oob.err), pch = 23, col = c("red", "blue"), type = "b", ylab="Mean Squared Error")legend("topright", legend = c("OOB", "Test"), pch = 23, col = c("red", "blue"))Idéalement, ces 2 courbes devraient s’aligner, mais il semble que l’erreur de test soit un peu plus faible. Cependant, il y a beaucoup de variabilité dans ces estimations d’erreur de test. Puisque l’estimation de l’erreur hors sac a été calculée sur un ensemble de données et que l’estimation de l’erreur de test a été calculée sur un autre ensemble de données, ces différences sont plutôt bien dans les erreurs standard.
Vous avez remarqué que la courbe rouge est doucement au-dessus de la courbe bleue ? Ces estimations d’erreurs sont très corrélées, car le randomForest avec mtry = 4 est très similaire à celui avec mtry = 5. C’est pourquoi chacune des courbes est assez lisse. Ce que vous voyez, c’est que mtry autour de 4 semble être le choix le plus optimal, au moins pour l’erreur de test. Cette valeur de mtry pour l’erreur hors-sac est égale à 9.
Donc avec très peu de paliers, vous avez ajusté un modèle de prédiction très puissant en utilisant les forêts aléatoires. Comment cela ? Le côté gauche montre la performance d’un seul arbre. L’erreur quadratique moyenne sur le hors-sac est de 26, et vous l’avez réduite à environ 15 (juste un peu plus de la moitié). Cela signifie que vous avez réduit l’erreur de moitié. De même pour l’erreur de test, vous avez réduit l’erreur de 20 à 12.
Boosting
Par rapport aux forêts aléatoires, le boosting fait pousser des arbres plus petits et plus tronqués et va au biais. Vous utiliserez le package GBM (Gradient Boosted Modeling), sous R.
require(gbm)GBM demande la distribution, qui est gaussienne, car vous ferez de la perte par erreur quadratique. Vous allez demander à GBM 10 000 arbres, ce qui semble beaucoup, mais il s’agira d’arbres peu profonds. La profondeur d’interaction est le nombre de splits, donc vous voulez 4 splits dans chaque arbre. Le rétrécissement est de 0,01, ce qui est combien vous allez rétrécir le pas d’arbre en arrière.
boost.boston = gbm(medv~., data = boston, distribution = "gaussian", n.trees = 10000, shrinkage = 0.01, interaction.depth = 4)summary(boost.boston)La fonction summary donne un tracé d’importance de variable. Il semble qu’il y ait 2 variables qui ont une grande importance relative : rm (nombre de pièces) et lstat (pourcentage de personnes de statut économique inférieur dans la communauté). Traçons ces 2 variables :
plot(boost.boston,i="lstat")plot(boost.boston,i="rm")Le 1er tracé montre que plus la proportion de personnes de statut économique inférieur dans la banlieue est élevée, plus la valeur des prix des logements est faible. Le 2e tracé montre la relation inverse avec le nombre de pièces : le nombre moyen de pièces dans la maison augmente lorsque le prix augmente.
Il est temps de prédire un modèle boosté sur le jeu de données de test. Regardons les performances du test en fonction du nombre d’arbres :
-
D’abord, vous faites une grille de nombre d’arbres par pas de 100 de 100 à 10 000.
-
Puis, vous exécutez la fonction
predictsur le modèle boosté. Elle prendn.treescomme argument, et produit une matrice de prédictions sur les données de test. -
Les dimensions de la matrice sont 206 observations de test et 100 vecteurs de prédiction différents aux 100 valeurs différentes de l’arbre.
n.trees = seq(from = 100, to = 10000, by = 100)predmat = predict(boost.boston, newdata = boston, n.trees = n.trees)dim(predmat)
Il est temps de calculer l’erreur de test pour chacun des vecteurs de prédiction:
-
predmatest une matrice,medvest un vecteur, donc (predmat–medv) est une matrice de différences. Vous pouvez utiliser la fonctionapplyaux colonnes de ces différences carrées (la moyenne). Cela permettrait de calculer l’erreur quadratique moyenne en colonne pour les vecteurs de prédiction. -
Puis vous faites un tracé en utilisant des paramètres similaires à celui utilisé pour Random Forest. Il s’agirait d’un graphique d’erreur de boosting.
boost.err = with(boston, apply( (predmat - medv)^2, 2, mean) )plot(n.trees, boost.err, pch = 23, ylab = "Mean Squared Error", xlab = "# Trees", main = "Boosting Test Error")abline(h = min(test.err), col = "red")
L’erreur de boosting diminue sensiblement lorsque le nombre d’arbres augmente. C’est une preuve montrant que le boosting est réticent à l’overfit. Incluons également la meilleure erreur de test de la randomForest dans le graphique. Le boosting obtient en fait une quantité raisonnable en dessous de l’erreur de test pour randomForest.
Conclusion
C’est donc la fin de ce tutoriel R sur la construction de modèles d’arbres de décision : arbres de classification, forêts aléatoires et arbres boostés. Les 2 dernières sont des méthodes puissantes que vous pouvez utiliser à tout moment selon vos besoins. D’après mon expérience, le boosting surpasse généralement RandomForest, mais RandomForest est plus facile à mettre en œuvre. Dans RandomForest, le seul paramètre de réglage est le nombre d’arbres ; tandis que dans boosting, plus de paramètres de réglage sont nécessaires en plus du nombre d’arbres, y compris le rétrécissement et la profondeur d’interaction.
Si vous souhaitez en savoir plus, assurez-vous de jeter un coup d’œil à notre cours Machine Learning Toolbox pour R.