Qu’est-ce que le PGS, dépistage génétique préimplantatoire ?
- Le PGS, dépistage génétique préimplantatoire, consiste à prélever une ou plusieurs cellules d’un embryon issu d’une fécondation in vitro afin de tester la normalité chromosomique.
- Le SGP permet de dépister chez l’embryon un nombre normal de chromosomes
- L’être humain possède 23 paires de chromosomes – pour un total de 46
- Avoir un chromosome supplémentaire ou manquant entraîne des problèmes
- Un exemple est le syndrome de Down qui présente un chromosome supplémentaire numéro 21. Cela devrait être détecté par le SGP.
- Le SGP ne teste pas une maladie spécifique telle que la fibrose kystique. Ce processus est appelé DPI (diagnostic génétique préimplantatoire).
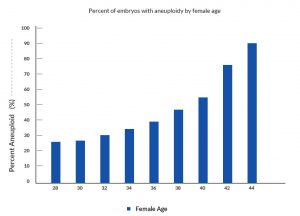
De nombreux embryons humains sont anormaux sur le plan chromosomique
Plusieurs études ont montré que, globalement, environ 50 % des embryons humains préimplantatoires issus de la FIV sont anormaux sur le plan chromosomique. Le taux d’anomalies est fortement affecté par l’âge de la femme, comme le montre le graphique ci-dessous. Les chromosomes des ovules de femmes plus âgées présentent un taux d’anomalies significativement plus élevé.
Que faire si la première tentative de FIV échoue
Des problèmes dans le passé avec le dépistage de l’aneuploïdie des embryons FIV
La FIV et le PGS pour l’aneuploïdie (un nombre anormal de chromosomes) ont été utilisés dans certaines cliniques aux États-Unis depuis le milieu des années 1990. Cependant, des études ont montré que la réalisation d’une biopsie d’embryon au troisième jour et la réalisation de l’analyse génétique à l’aide de la technologie FISH (hybridation in situ fluorescente) n’entraînaient pas une augmentation des chances pour le patient d’avoir un cycle de FIV réussi.
Il y avait 2 problèmes principaux avec cette approche.
Souvent, après l’échec répété de l’implantation de tout embryon avec la FIV, les femmes diront;
- La technologie FISH ne regardait généralement que 5 chromosomes sur 23. Par conséquent, le test FISH manquait de nombreuses anomalies chromosomiques. Il en résultait que des embryons anormaux étaient transférés après que le test de dépistage soit revenu « normal ».
- Les biopsies du jour 3 consistaient à retirer une cellule (ou 2 cellules) d’un embryon de 6 à 10 cellules. Cela nécessitait de faire un trou relativement grand dans l’enveloppe de l’embryon, puis de retirer un pourcentage important de la « biomasse » de cet embryon (un sixième à un dixième retiré, voire plus).
Des progrès récents permettent de meilleurs taux de réussite en FIV après un dépistage d’aneuploïdie
Les progrès dans ce domaine ont conduit à l’utilisation de technologies génétiques améliorées qui permettent d’évaluer les 23 paires de chromosomes.
Il existe actuellement 4 technologies qui peuvent être utilisées pour l’évaluation de la normalité de l’ensemble des 23 chromosomes :
- Séquençage de nouvelle génération (NGS)
- Hybridation génomique comparative en réseau (aCGH)
- Micro-réseaux de polymorphisme mononucléotidique (SNP)
- Réaction en chaîne par polymérase quantitative en temps réel (qPCR)
L’hybridation génomique comparative (souvent appelée CGH, ou aCGH) est une technologie de microarray qui est souvent utilisée maintenant à la place de la FISH, plus ancienne et beaucoup moins complète. Avec la CGH par microréseau, l’ADN réel de l’embryon est comparé à un échantillon d’ADN normal connu en utilisant des milliers de marqueurs génétiques spécifiques. Cela donne un résultat plus précis, avec beaucoup moins de faux résultats normaux ou de faux résultats anormaux.
Certaines études ont déterminé que le taux d’erreur en utilisant la technologie CGH en réseau est d’environ 2%. La FISH a un taux d’erreur d’environ 5 à 10 %. En outre, de nombreux autres embryons anormaux seraient signalés par la FISH comme normaux parce que l’anomalie se trouvait dans un chromosome qui ne faisait pas partie du panel FISH utilisé.
Le séquençage de nouvelle génération (NGS) est une technologie plus récente qui est de plus en plus utilisée pour tester les embryons de FIV depuis environ 2015. Le NGS semble être meilleur pour détecter les changements segmentaires plus petits par rapport à l’aCGH. On pense également qu’il est meilleur pour détecter les aneuploïdies partielles et les petites translocations non équilibrées. Le mosaïcisme est probablement plus susceptible d’être découvert en utilisant le séquençage de nouvelle génération.
Améliorations des techniques de biopsie embryonnaire
La biopsie durophectoderme est effectuée au stade du blastocyste, au jour 5 et 6. A ce stade, il y a beaucoup plus de cellules présentes dans l’embryon. Cela permet de prélever de multiples cellules du trophectoderme (précurseurs du placenta). Les cellules de la masse interne (précurseurs du fœtus) peuvent être laissées intactes pendant la biopsie.
Lors de la biopsie du trophectoderme, environ 5 cellules sont coupées pour le test génétique. Cela n’affaiblit pas significativement l’embryon car il possède environ 70 à 150 cellules à ce stade.
La combinaison de ces deux modifications (génétique avancée et biopsie de trophectoderme) a permis d’améliorer significativement les taux de réussite de grossesse pour les patients qui veulent utiliser le PGS pour leur traitement de FIV.
Certaines cliniques aux États-Unis ont utilisé la biopsie de trophectoderme et les nouvelles technologies de génétique pour dépister les embryons chez certains patients en FIV. Les résultats observés dans certains programmes de FIV (y compris les nôtres) ont été très prometteurs.
- Nous constatons une amélioration substantielle des taux de grossesse en cours chez les patients qui subissent une biopsie du trophectoderme au stade du blastocyste avec une congélation ultérieure de leurs embryons.
- Un cycle de transfert congelé-décongelé est effectué après le retour des résultats de l’analyse chromosomique
Problèmes de réceptivité utérine
Il existe des spéculations intéressantes selon lesquelles la muqueuse utérine pourrait être moins réceptive pendant un cycle stimulé par rapport au cycle de remplacement d’embryon contrôlé ou « artificiel ».
- Certains médecins de la fertilité croient que le transfert d’embryons dans un cycle contrôlé (en utilisant des embryons congelés) donne un taux de grossesse plus élevé que dans un cycle « frais »
- Cela n’a pas encore été soigneusement étudié avec de multiples essais cliniques contrôlés
- L’amélioration du taux de réussite observée après une biopsie de blastocyste et une analyse chromosomique complète est principalement due au bénéfice du transfert d’embryons chromosomiquement normaux
- Chez certaines femmes, il existe un bénéfice supplémentaire dérivé du transfert du ou des embryons dans un cycle congelé-décongelé plutôt que dans un cycle stimulé
À quels couples devrions-nous proposer le dépistage des aneuploïdies ?
- C’est actuellement une question en évolution dans le domaine de la médecine reproductive
- Certains candidats potentiels pourraient être :
- Les femmes de plus de (environ) 35 ans qui font de la FIV
- Des patients de tout âge qui ont échoué plusieurs cycles de FIV. Ils veulent des réponses sur les raisons de leur échec. Ils veulent également savoir ce qu’ils doivent faire pour améliorer leurs chances d’avoir un bébé. Le PGS fournit parfois des réponses.
- Le PGS peut également permettre un « désherbage » des embryons anormaux. Par exemple, si un embryon sur 6 est chromosomiquement normal et que 5 sur 6 sont anormaux – on transfère l’embryon normal et on devrait avoir de très bonnes chances d’avoir un bébé.
- Couples avec des fausses couches récurrentes
- Toute personne qui veut utiliser cette technologie pour dépister ses embryons afin d’en transférer un qui est testé comme chromosomiquement normal – et qui a donc de grandes chances de s’implanter et de faire un bébé
Coût du DPI – SGP
Un autre problème est le coût. Les coûts du DPI aux États-Unis varient d’environ 3000 à 9000 $, plus tous les autres coûts associés à la FIV. Il y a des coûts liés au DPI associés à la procédure de biopsie d’embryon elle-même et il y a aussi des coûts impliqués pour le laboratoire de génétique qui effectue l’analyse chromosomique sur les cellules.
- Pour faire un DPI, il faut d’abord faire une FIV (et payer) les coûts standard de la FIV
- Puis il y a le coût de la biopsie et l’analyse génétique des cellules
- Il peut y avoir des frais supplémentaires pour les embryons congelés. peut y avoir des frais supplémentaires pour le cycle de transfert d’embryons congelés qui est effectué après les résultats de la génétique
Coûts du DPI dans notre centre
Quelles sont les meilleures cliniques de DPI ?
En général, les meilleures cliniques pour le DPI ou le PGS seront celles qui ont les meilleurs taux de réussite en FIV. Il est essentiel d’avoir un excellent système de culture afin d’obtenir un nombre maximal de blastocystes de qualité pour la biopsie.
De plus, les compétences qui conduisent à des taux de réussite élevés en FIV sont les mêmes que celles qui facilitent la réalisation de la culture des blastocystes, la biopsie du trophectoderme, la vitrification (congélation) des blastocystes et la réussite des cycles de transfert congelés-décongelés.
- Vérifiez les sites du CDC et du SART pour trouver une clinique de FIV dans votre région avec un bon volume de cas et des taux de réussite élevés.
- Vérifiez qu’ils ont fait des biopsies de trophectoderme
- Ou, venez simplement à notre clinique pour la FIV et le DPI
.