Introduzione
L’albero decisionale è un tipo di algoritmo di apprendimento supervisionato che può essere usato in problemi di regressione e classificazione. Funziona per variabili di input e di output sia categoriche che continue.
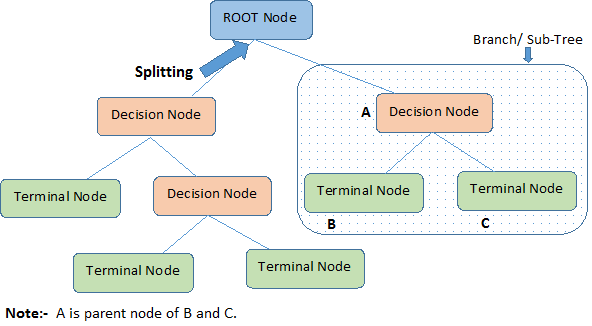
Identifichiamo le terminologie importanti dell’albero di decisione, guardando l’immagine sopra:
-
Il nodo radice rappresenta l’intera popolazione o campione. Viene ulteriormente diviso in due o più insiemi omogenei.
-
Splitting è un processo di divisione di un nodo in due o più sotto-nodi.
-
Quando un sotto-nodo si divide in ulteriori sotto-nodi, è chiamato Nodo di decisione.
-
I nodi che non si dividono sono chiamati Nodo Terminale o Foglia.
-
Quando si rimuovono i sotto-nodi di un nodo di decisione, questo processo è chiamato Pruning. L’opposto della potatura è lo Splitting.
-
Una sottosezione di un intero albero è chiamata Branch.
-
Un nodo, che è diviso in sotto-nodi è chiamato nodo padre dei sotto-nodi; mentre i sotto-nodi sono chiamati figli del nodo padre.
Tipi di alberi di decisione
Alberi di regressione
Diamo un’occhiata all’immagine sottostante, che aiuta a visualizzare la natura del partizionamento effettuato da un albero di regressione. Questo mostra un albero non potato e un albero di regressione adattato a un set di dati casuale. Entrambe le visualizzazioni mostrano una serie di regole di divisione, a partire dalla cima dell’albero. Notate che ogni divisione del dominio è allineata con uno degli assi delle caratteristiche. Il concetto di divisione parallela degli assi si generalizza direttamente a dimensioni maggiori di due. Per uno spazio di caratteristiche di dimensione $p$, un sottoinsieme di $\mathbb{R}^p$, lo spazio è diviso in $M$ regioni, $R_{m}$, ognuna delle quali è un “iperblocco” $p$-dimensionale.
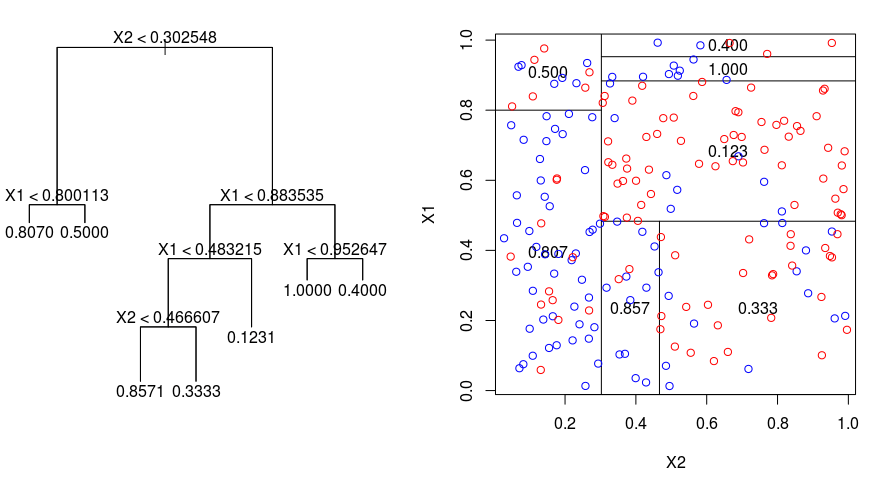
Per costruire un albero di regressione, si usa prima la divisione binaria ricorsiva per far crescere un grande albero sui dati di allenamento, fermandosi solo quando ogni nodo terminale ha meno di un numero minimo di osservazioni. Il Recursive Binary Splitting è un algoritmo greedy e top-down usato per minimizzare la Somma Residua dei Quadri (RSS), una misura di errore usata anche nelle impostazioni di regressione lineare. La RSS, nel caso di uno spazio delle caratteristiche partizionato con M partizioni è data da:
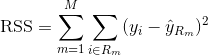
Partendo dalla cima dell’albero, lo si divide in 2 rami, creando una partizione di 2 spazi. Poi si esegue questa particolare divisione in cima all’albero più volte e si sceglie la divisione delle caratteristiche che minimizza l’RSS (attuale).
In seguito, si applica la potatura della complessità dei costi al grande albero per ottenere una sequenza dei migliori sottoalberi, in funzione di $\alpha$. L’idea di base qui è di introdurre un ulteriore parametro di regolazione, denotato da $\alpha$ che bilancia la profondità dell’albero e la sua bontà di adattamento ai dati di formazione.
Puoi usare la cross-validazione K-fold per scegliere $\alpha$. Questa tecnica consiste semplicemente nel dividere le osservazioni di allenamento in K pieghe per stimare il tasso di errore di test dei sottoalberi. Il tuo obiettivo è quello di selezionare quello che porta al più basso tasso di errore.
Alberi di classificazione
Un albero di classificazione è molto simile a un albero di regressione, tranne che viene utilizzato per prevedere una risposta qualitativa piuttosto che una quantitativa.
Ricorda che per un albero di regressione, la risposta prevista per un’osservazione è data dalla risposta media delle osservazioni di allenamento che appartengono allo stesso nodo terminale. Al contrario, per un albero di classificazione, si prevede che ogni osservazione appartenga alla classe più comunemente presente tra le osservazioni di allenamento nella regione a cui appartiene.
Nell’interpretare i risultati di un albero di classificazione, si è spesso interessati non solo alla previsione della classe corrispondente a una particolare regione del nodo terminale, ma anche alle proporzioni delle classi tra le osservazioni di allenamento che cadono in quella regione.
Il compito di far crescere un albero di classificazione è abbastanza simile a quello di un albero di regressione. Proprio come nella regressione, si usa la divisione binaria ricorsiva per far crescere un albero di classificazione. Tuttavia, nell’impostazione di classificazione, la somma residua dei quadrati non può essere utilizzata come criterio per effettuare le suddivisioni binarie. Invece, puoi usare uno dei 3 metodi seguenti:
- Classification Error Rate: Piuttosto che vedere quanto una risposta numerica è lontana dal valore medio, come nell’impostazione della regressione, si può invece definire il “tasso di errore” come la frazione di osservazioni di allenamento in una particolare regione che non appartengono alla classe più diffusa. L’errore è dato da questa equazione:
E = 1 – argmaxc(${hat{\pi}_{mc}$)
in cui ${hat{\pi}_{mc}$ rappresenta la frazione di dati di allenamento nella regione Rm che appartiene alla classe c.
- Indice di Gini: L’indice di Gini è una metrica di errore alternativa che è progettata per mostrare quanto sia “pura” una regione. “Purezza” in questo caso significa quanta parte dei dati di allenamento in una particolare regione appartiene ad una singola classe. Se una regione Rm contiene dati che provengono principalmente da una singola classe c, allora il valore dell’indice Gini sarà piccolo:
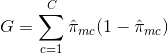
- Cross-Entropia: Una terza alternativa, che è simile all’indice di Gini, è conosciuta come la Cross-Entropia o Devianza:
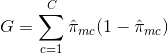
La cross-entropia assumerà un valore vicino allo zero se i $hat{\pi}_{mc}$ sono tutti vicini a 0 o vicini a 1. Quindi, come l’indice di Gini, l’entropia incrociata assumerà un valore piccolo se il mesimo nodo è puro. Infatti, si scopre che l’indice di Gini e l’entropia incrociata sono numericamente molto simili.
Quando si costruisce un albero di classificazione, l’indice di Gini o l’entropia incrociata sono tipicamente usati per valutare la qualità di una particolare divisione, poiché sono più sensibili alla purezza dei nodi di quanto non lo sia il tasso di errore di classificazione. Ognuno di questi 3 approcci può essere usato quando si pota l’albero, ma il tasso di errore di classificazione è preferibile se l’accuratezza di previsione dell’albero finale potato è l’obiettivo.
Svantaggi e svantaggi degli alberi decisionali
Il principale vantaggio di usare gli alberi decisionali è che sono intuitivamente molto facili da spiegare. Rispecchiano da vicino il processo decisionale umano rispetto ad altri approcci di regressione e classificazione. Possono essere visualizzati graficamente, e possono facilmente gestire predittori qualitativi senza la necessità di creare variabili dummy.
Tuttavia, gli alberi decisionali generalmente non hanno lo stesso livello di accuratezza predittiva di altri approcci, poiché non sono abbastanza robusti. Un piccolo cambiamento nei dati può causare un grande cambiamento nell’albero finale stimato.
Aggregando molti alberi decisionali, usando metodi come bagging, foreste casuali e boosting, la performance predittiva degli alberi decisionali può essere sostanzialmente migliorata.
Metodi basati sugli alberi
Bagging
Gli alberi decisionali discussi sopra soffrono di un’alta varianza, il che significa che se si dividono i dati di allenamento in 2 parti a caso, e si adatta un albero decisionale ad entrambe le metà, i risultati che si ottengono potrebbero essere molto diversi. Al contrario, una procedura con bassa varianza darà risultati simili se applicata ripetutamente a set di dati distinti.
Bagging, o aggregazione bootstrap, è una tecnica usata per ridurre la varianza delle vostre previsioni combinando il risultato di più classificatori modellati su diversi sottocampioni dello stesso set di dati. Ecco l’equazione per il bagging:

in cui si generano $B$ diversi set di dati di allenamento bootstrap. Poi addestrate il vostro metodo sul $bth$ set di addestramento bootstrapped per ottenere $\hat{f}_{b}(x)$, e infine fate la media delle previsioni.
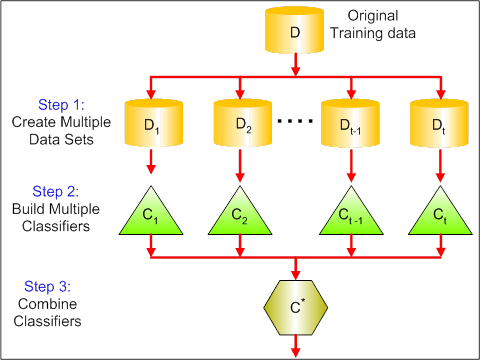
La figura qui sotto mostra i 3 diversi passi del bagging:
-
Step 1: Qui si sostituiscono i dati originali con nuovi dati. I nuovi dati di solito hanno una frazione delle colonne e delle righe dei dati originali, che poi possono essere usati come iper-parametri nel modello di bagging.
-
Step 2: Si costruiscono i classificatori su ogni set di dati. Generalmente, si può usare lo stesso classificatore per fare modelli e previsioni.
-
Step 3: Infine, si usa un valore medio per combinare le previsioni di tutti i classificatori, a seconda del problema. Generalmente, questi valori combinati sono più robusti di un singolo modello.
Mentre il bagging può migliorare le previsioni per molti metodi di regressione e classificazione, è particolarmente utile per gli alberi di decisione. Per applicare il bagging agli alberi di regressione/classificazione, si costruiscono semplicemente $B$ alberi di regressione/classificazione usando $B$ insiemi di training bootstrapped, e si fa la media delle previsioni risultanti. Questi alberi sono cresciuti in profondità e non vengono potati. Quindi ogni singolo albero ha un’alta varianza, ma un basso bias. La media di questi $B$ alberi riduce la varianza.
In senso lato, è stato dimostrato che il bagging dà miglioramenti impressionanti nella precisione combinando insieme centinaia o anche migliaia di alberi in una singola procedura.
Foreste casuali
Le foreste casuali sono un metodo versatile di apprendimento automatico in grado di eseguire sia compiti di regressione che di classificazione. Intraprende anche metodi di riduzione dimensionale, tratta i valori mancanti, i valori outlier e altri passi essenziali dell’esplorazione dei dati, e fa un lavoro abbastanza buono.
Random Forests fornisce un miglioramento rispetto agli alberi insaccati con una piccola modifica che decorre gli alberi. Come nel bagging, si costruisce un certo numero di alberi decisionali su campioni di allenamento bootstrapped. Ma quando si costruiscono questi alberi decisionali, ogni volta che si considera una divisione in un albero, un campione casuale di m predittori viene scelto come candidato alla divisione dall’insieme completo di $p$ predittori. La divisione può utilizzare solo uno di questi $m$ predittori. Questa è la differenza principale tra le foreste casuali e il bagging; perché come nel bagging, la scelta del predittore $m = p$.
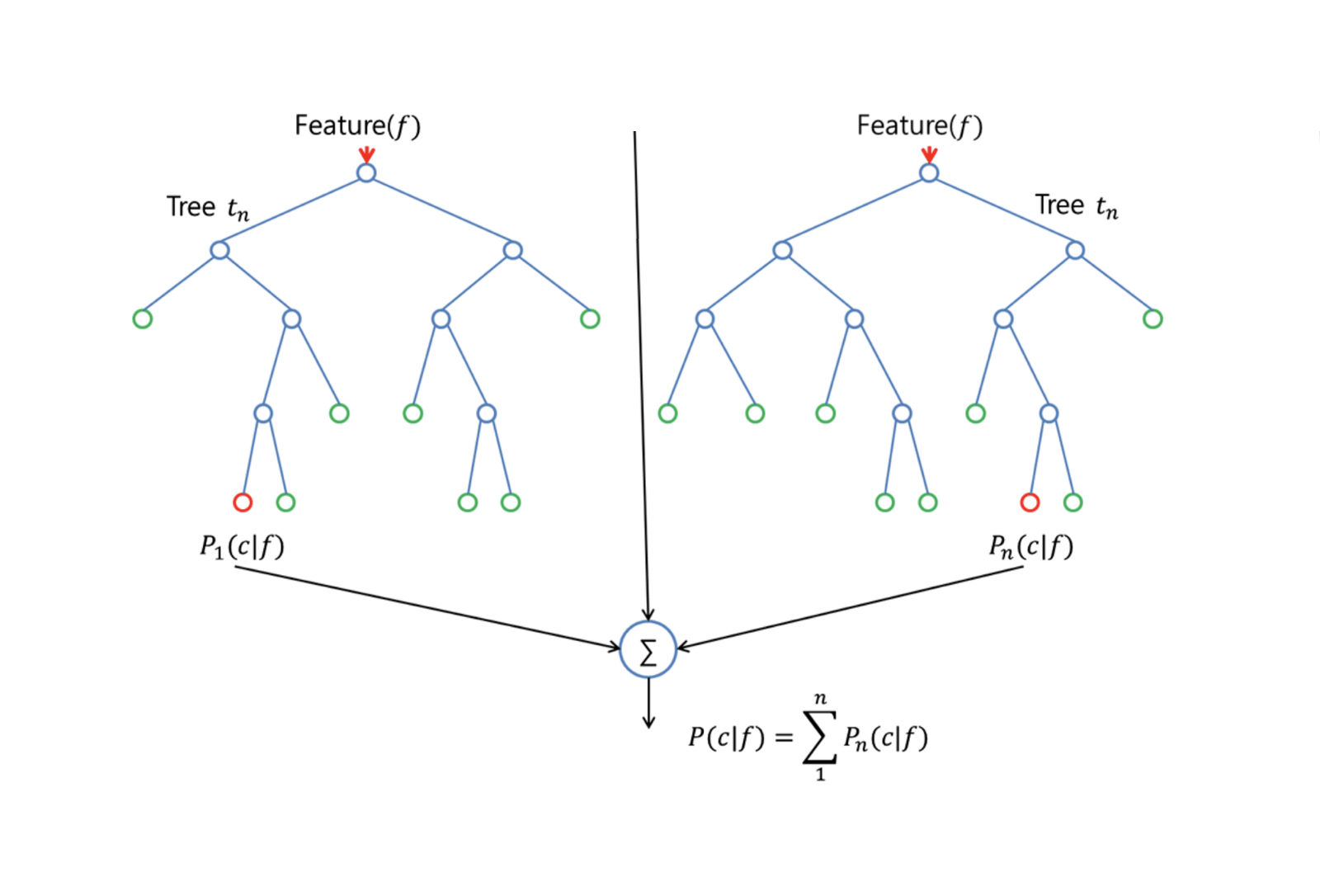
Per far crescere una foresta casuale, si dovrebbe:
-
Prima supporre che il numero di casi nel set di allenamento sia K. Poi, prendi un campione casuale di questi K casi, e poi usa questo campione come set di allenamento per far crescere l’albero.
-
Se ci sono $p$ variabili di input, specifica un numero $m < p$ tale che ad ogni nodo, puoi selezionare $m$ variabili casuali tra le $p$. La migliore divisione su queste $m$ viene usata per dividere il nodo.
-
Ogni albero viene successivamente fatto crescere il più possibile e non è necessaria alcuna potatura.
-
Infine, aggregare le previsioni degli alberi di destinazione per prevedere nuovi dati.
Random Forests è molto efficace nella stima dei dati mancanti e nel mantenere la precisione quando manca una grande proporzione di dati. Può anche bilanciare gli errori in serie di dati in cui le classi sono sbilanciate. Soprattutto, può gestire serie di dati massicce con grande dimensionalità. Tuttavia, uno svantaggio dell’uso delle Foreste casuali è che si potrebbe facilmente sovradimensionare gli insiemi di dati rumorosi, specialmente nel caso della regressione.
Boosting
Boosting è un altro approccio per migliorare le previsioni risultanti da un albero decisionale. Come il bagging e le foreste casuali, è un approccio generale che può essere applicato a molti metodi di apprendimento statistico per la regressione o la classificazione. Ricordiamo che il bagging implica la creazione di più copie del dataset di allenamento originale usando il bootstrap, l’adattamento di un albero decisionale separato ad ogni copia, e poi la combinazione di tutti gli alberi per creare un unico modello predittivo. In particolare, ogni albero è costruito su un set di dati bootstrap, indipendente dagli altri alberi.
Il Boosting funziona in modo simile, tranne che gli alberi sono cresciuti in modo sequenziale: ogni albero è cresciuto utilizzando le informazioni degli alberi cresciuti in precedenza. Il Boosting non comporta un campionamento bootstrap; invece, ogni albero è montato su una versione modificata del dataset originale.

Per entrambi gli alberi di regressione e classificazione, il boosting funziona così:
-
A differenza dell’adattamento di un singolo grande albero decisionale ai dati, che equivale ad adattare i dati duramente e potenzialmente in eccesso, l’approccio boosting invece impara lentamente.
-
Dato il modello corrente, si adatta un albero decisionale ai residui del modello. Cioè, si adatta un albero usando i residui attuali, piuttosto che il risultato $Y$, come risposta.
-
Si aggiunge poi questo nuovo albero decisionale nella funzione adattata per aggiornare i residui. Ognuno di questi alberi può essere piuttosto piccolo, con pochi nodi terminali, determinati dal parametro $d$ nell’algoritmo. Adattando piccoli alberi ai residui, si migliora lentamente $\hat{f}$ nelle aree in cui non funziona bene.
-
Il parametro di restringimento $\nu$ rallenta ulteriormente il processo, permettendo a più alberi di forma diversa di attaccare i residui.
Il Boosting è molto utile quando si hanno molti dati e ci si aspetta che gli alberi di decisione siano molto complessi. Il boosting è stato usato per risolvere molti problemi di classificazione e regressione, tra cui l’analisi del rischio, l’analisi del sentimento, la pubblicità predittiva, la modellazione dei prezzi, la stima delle vendite e la diagnosi dei pazienti, tra gli altri.
Alberi di decisione in R
Alberi di classificazione
Per questa parte, si lavora con il dataset Carseats usando il pacchetto tree in R. Ricordate che è necessario installare prima i pacchetti ISLR e tree nel vostro ambiente R Studio. Carichiamo prima il dataframe Carseats dal pacchetto ISLR.
library(ISLR)data(package="ISLR")carseats<-CarseatsCarichiamo anche il pacchetto tree.
require(tree)Il dataset Carseats è un dataframe con 400 osservazioni sulle seguenti 11 variabili:
-
Vendite: vendite unitarie in migliaia
-
CompPrice: prezzo praticato dal concorrente in ogni luogo
-
Income: livello di reddito della comunità in migliaia di dollari
-
Pubblicità: budget pubblicitario locale in ogni località in migliaia di dollari
-
Popolazione: popolazione regionale in migliaia
-
Prezzo: prezzo dei posti auto in ogni località
-
ShelveLoc: Bad, Good o Medium indica la qualità della posizione degli scaffali
-
Age: livello di età della popolazione
-
Education: livello di istruzione nel luogo
-
Urban: Sì/No
-
USA: Sì/No
names(carseats)Diamo un’occhiata all’istogramma delle vendite di automobili:
hist(carseats$Sales)Osservate che Sales è una variabile quantitativa. Volete dimostrarlo usando alberi con una risposta binaria. Per farlo, si trasforma Sales in una variabile binaria, che si chiamerà High. Se il fatturato è inferiore a 8, non sarà alto. Altrimenti, sarà alto. Poi potete rimettere la nuova variabile High nel dataframe.
High = ifelse(carseats$Sales<=8, "No", "Yes")carseats = data.frame(carseats, High)Ora riempiamo un modello usando alberi decisionali. Naturalmente, non potete avere la variabile Sales qui perché la vostra variabile di risposta High è stata creata da Sales. Quindi, escludiamola e adattiamo l’albero.
tree.carseats = tree(High~.-Sales, data=carseats)Vediamo il riassunto del tuo albero di classificazione:
summary(tree.carseats)Puoi vedere le variabili coinvolte, il numero di nodi terminali, la devianza media residua, così come il tasso di errore di classificazione. Per renderlo più visivo, tracciamo anche l’albero, poi annotiamolo usando la comoda funzione text:
plot(tree.carseats)text(tree.carseats, pretty = 0)Ci sono così tante variabili che è molto complicato guardare l’albero. Per lo meno, potete vedere che ad ogni nodo terminale, sono etichettati Yes o No. Ad ogni nodo di divisione, le variabili e il valore della scelta di divisione sono mostrati (per esempio, Price < 92.5 o Advertising < 13.5).
Per un riassunto dettagliato dell’albero, basta stamparlo. Sarà utile se vuoi estrarre dettagli dall’albero per altri scopi:
tree.carseatsÈ ora di potare l’albero. Creiamo un set di allenamento e un test dividendo il dataframe carseats in 250 campioni di allenamento e 150 campioni di test. Per prima cosa, si imposta un seme per rendere i risultati riproducibili. Poi si prende un campione casuale dei numeri ID (indice) dei campioni. In particolare qui, si campiona dall’insieme da 1 a n numero di file di posti auto, che è 400. Volete un campione di dimensione 250 (per default, il campione usa senza sostituzione).
set.seed(101)train=sample(1:nrow(carseats), 250)Quindi ora ottenete questo indice di train, che indicizza 250 delle 400 osservazioni. Potete reinserire il modello con tree, usando la stessa formula, ma dicendo all’albero di usare un sottoinsieme uguale a train. Poi facciamo un grafico:
tree.carseats = tree(High~.-Sales, carseats, subset=train)plot(tree.carseats)text(tree.carseats, pretty=0)Il grafico appare un po’ diverso a causa del set di dati leggermente diverso. Tuttavia, la complessità dell’albero sembra più o meno la stessa.
Ora prendiamo questo albero e lo prevediamo sul set di test, usando il metodo predict per gli alberi. Qui vorrete effettivamente predire le class etichette.
tree.pred = predict(tree.carseats, carseats, type="class")Poi potrete valutare l’errore usando una tabella di misclassificazione.
with(carseats, table(tree.pred, High))Sulle diagonali ci sono le classificazioni corrette, mentre fuori dalle diagonali ci sono quelle errate. Volete recensire solo quelle corrette. Per farlo, si può prendere la somma delle 2 diagonali divisa per il totale (150 osservazioni di prova).
(72 + 43) / 150Ok, si ottiene un errore di 0,76 con questo albero.
Quando si cresce un grande albero cespuglioso, potrebbe avere troppa varianza. Quindi, usiamo la validazione incrociata per potare l’albero in modo ottimale. Usando cv.tree, userete l’errore di classificazione come base per fare la potatura.
cv.carseats = cv.tree(tree.carseats, FUN = prune.misclass)cv.carseatsLa stampa dei risultati mostra i dettagli del percorso della validazione incrociata. Potete vedere le dimensioni degli alberi man mano che sono stati potati indietro, le deviazioni man mano che la potatura procedeva, così come il parametro di complessità dei costi usato nel processo.
Stampiamo questo:
plot(cv.carseats)Guardando il grafico, si vede una parte di spirale verso il basso a causa dell’errore di classificazione su 250 punti convalidati. Quindi scegliamo un valore nella parte discendente (12). Poi, potiamo l’albero ad una dimensione di 12 per identificare quell’albero. Infine, tracciamo e annotiamo quell’albero per vedere il risultato.
prune.carseats = prune.misclass(tree.carseats, best = 12)plot(prune.carseats)text(prune.carseats, pretty=0)È un po’ meno profondo degli alberi precedenti, e si possono effettivamente leggere le etichette. Valutiamolo di nuovo sul set di dati di test.
tree.pred = predict(prune.carseats, carseats, type="class")with(carseats, table(tree.pred, High))(74 + 39) / 150Sembra che le classificazioni corrette siano calate un po’. Ha fatto circa lo stesso del tuo albero originale, quindi la potatura non ha fatto molto male rispetto agli errori di classificazione, e ha dato un albero più semplice.
Spesso gli alberi non danno errori di predizione molto buoni, quindi diamo un’occhiata alle foreste casuali e al boosting, che tendono a superare gli alberi per quanto riguarda la predizione e gli errori di classificazione.
Foreste casuali
Per questa parte, userete il Boston housing data per esplorare le foreste casuali e il boosting. Il set di dati si trova nel pacchetto MASS. Fornisce i valori delle abitazioni e altre statistiche in ciascuno dei 506 sobborghi di Boston basati su un censimento del 1970.
library(MASS)data(package="MASS")boston<-Bostondim(boston)names(boston)Carichiamo anche il pacchetto randomForest.
require(randomForest)Per preparare i dati per la foresta casuale, impostiamo il seme e creiamo un training set campione di 300 osservazioni.
set.seed(101)train = sample(1:nrow(boston), 300)In questo dataset, ci sono 506 periferie di Boston. Per ogni periferia, si hanno variabili come la criminalità pro capite, i tipi di industria, il numero medio di stanze per abitazione, la percentuale media di età delle case, ecc. Usiamo medv – il valore mediano delle case occupate dai proprietari per ciascuno di questi sobborghi, come variabile di risposta.
Adattiamo una foresta casuale e vediamo come si comporta. Come detto, si usa la risposta medv, il valore mediano delle abitazioni (in $1K dollari), e il set di campioni di allenamento.
rf.boston = randomForest(medv~., data = boston, subset = train)rf.bostonStampando la foresta casuale si ottiene il suo riassunto: il numero di alberi (ne sono stati coltivati 500), i residui quadrati medi (MSR), e la percentuale di varianza spiegata. L’MSR e la % di varianza spiegata si basano sulle stime out-of-bag, un espediente molto intelligente nelle foreste casuali per ottenere stime di errore oneste.
L’unico parametro di tuning in una Foresta casuale è l’argomento chiamato mtry, che è il numero di variabili che vengono selezionate ad ogni split di ogni albero quando si fa uno split. Come si vede qui, mtry è 4 delle 13 variabili esplorative (escluso medv) nei dati di Boston Housing – il che significa che ogni volta che l’albero arriva a dividere un nodo, 4 variabili verrebbero selezionate a caso, poi la divisione sarebbe limitata a 1 di quelle 4 variabili. Questo è il modo in cui randomForests de-correla gli alberi.
Si sta per adattare una serie di foreste casuali. Ci sono 13 variabili, quindi facciamo in modo che mtry vada da 1 a 13:
-
Per registrare gli errori, si impostano 2 variabili
oob.erretest.err. -
In un ciclo di
mtryda 1 a 13, si adatta prima larandomForestcon quel valore dimtrysul datasettrain, limitando il numero di alberi a 350. -
Poi si estrae l’errore quadratico medio sull’oggetto (l’errore out-of-bag).
-
Poi si predice sul dataset di test (
boston) usandofit(il fit dirandomForest). -
Infine, si calcola l’errore del test: l’errore quadratico medio, che è uguale a
mean( (medv - pred) ^ 2 ).
oob.err = double(13)test.err = double(13)for(mtry in 1:13){ fit = randomForest(medv~., data = boston, subset=train, mtry=mtry, ntree = 350) oob.err = fit$mse pred = predict(fit, boston) test.err = with(boston, mean( (medv-pred)^2 ))}In pratica avete appena fatto crescere 4550 alberi (13 volte 350). Ora facciamo un grafico usando il comando matplot. L’errore di prova e l’errore fuori sacco sono legati insieme per fare una matrice a 2 colonne. Ci sono alcuni altri argomenti nella matrice, inclusi i valori dei caratteri di tracciatura (pch = 23 significa diamante riempito), i colori (rosso e blu), tipo uguale a entrambi (tracciando entrambi i punti e collegandoli con le linee), e il nome dell’asse y (Mean Squared Error). Puoi anche mettere una legenda nell’angolo in alto a destra del grafico.
matplot(1:mtry, cbind(test.err, oob.err), pch = 23, col = c("red", "blue"), type = "b", ylab="Mean Squared Error")legend("topright", legend = c("OOB", "Test"), pch = 23, col = c("red", "blue"))Idealmente, queste 2 curve dovrebbero allinearsi, ma sembra che l’errore del test sia un po’ più basso. Tuttavia, c’è molta variabilità in queste stime di errore di test. Poiché la stima dell’errore fuori dal sacco è stata calcolata su un set di dati e la stima dell’errore di test è stata calcolata su un altro set di dati, queste differenze sono abbastanza ben all’interno degli errori standard.
Nota che la curva rossa è leggermente sopra la curva blu? Queste stime di errore sono molto correlate, perché il randomForest con mtry = 4 è molto simile a quello con mtry = 5. Ecco perché ognuna delle curve è abbastanza liscia. Quello che si vede è che mtry intorno a 4 sembra essere la scelta più ottimale, almeno per l’errore di test. Questo valore di mtry per l’errore fuori sacco è uguale a 9.
Quindi, con pochissimi livelli, avete montato un modello di predizione molto potente usando le foreste casuali. In che modo? Il lato sinistro mostra la performance di un singolo albero. L’errore quadratico medio su out-of-bag è 26, e tu sei sceso a circa 15 (poco più della metà). Questo significa che hai ridotto l’errore della metà. Allo stesso modo per l’errore di test, hai ridotto l’errore da 20 a 12.
Boosting
Rispetto alle foreste casuali, il boosting fa crescere alberi più piccoli e stopposi e va al bias. Userete il pacchetto GBM (Gradient Boosted Modeling), in R.
require(gbm)GBM chiede la distribuzione, che è gaussiana, perché farete la perdita per errore quadratico. Chiederete a GBM 10.000 alberi, che sembrano molti, ma saranno alberi poco profondi. La profondità di interazione è il numero di spaccature, quindi volete 4 spaccature in ogni albero. Shrinkage è 0.01, che è quanto si restringe il passo dell’albero.
boost.boston = gbm(medv~., data = boston, distribution = "gaussian", n.trees = 10000, shrinkage = 0.01, interaction.depth = 4)summary(boost.boston)La funzione summary dà un grafico dell’importanza delle variabili. Sembra che ci siano 2 variabili che hanno un’alta importanza relativa: rm (numero di stanze) e lstat (percentuale di persone di status economico inferiore nella comunità). Tracciamo queste 2 variabili:
plot(boost.boston,i="lstat")plot(boost.boston,i="rm")Il primo grafico mostra che più alta è la percentuale di persone di status economico inferiore nel sobborgo, più basso è il valore dei prezzi delle abitazioni. Il 2° grafico mostra la relazione inversa con il numero di stanze: il numero medio di stanze nella casa aumenta all’aumentare del prezzo.
È il momento di prevedere un modello boosted sul dataset di test. Guardiamo la performance del test in funzione del numero di alberi:
-
Prima, si fa una griglia di numero di alberi a passi di 100 da 100 a 10.000.
-
Poi, si esegue la funzione
predictsul modello boosted. Prenden.treescome argomento e produce una matrice di predizioni sui dati di test. -
Le dimensioni della matrice sono 206 osservazioni di test e 100 diversi vettori di predizione ai 100 diversi valori di albero.
n.trees = seq(from = 100, to = 10000, by = 100)predmat = predict(boost.boston, newdata = boston, n.trees = n.trees)dim(predmat)
È il momento di calcolare l’errore di test per ciascuno dei vettori di previsione:
-
predmatè una matrice,medvè un vettore, quindi (predmat–medv) è una matrice di differenze. Potete usare la funzioneapplyper le colonne di queste differenze quadrate (la media). Questo calcolerebbe l’errore quadratico medio in colonna per i vettori di previsione. -
Poi fai un grafico usando parametri simili a quello usato per Random Forest. Mostrerebbe un grafico dell’errore di boosting.
boost.err = with(boston, apply( (predmat - medv)^2, 2, mean) )plot(n.trees, boost.err, pch = 23, ylab = "Mean Squared Error", xlab = "# Trees", main = "Boosting Test Error")abline(h = min(test.err), col = "red")
L’errore di boosting diminuisce all’aumentare del numero di alberi. Questa è una prova che dimostra che il boosting è riluttante all’overfit. Includiamo anche il miglior errore di test del randomForest nel grafico. Boosting effettivamente ottiene una quantità ragionevole al di sotto dell’errore di test per randomForest.
Conclusione
Così è la fine di questo tutorial R sulla costruzione di modelli di alberi decisionali: alberi di classificazione, foreste casuali e alberi boosted. Gli ultimi 2 sono metodi potenti che potete usare in qualsiasi momento se necessario. Nella mia esperienza, il boosting di solito supera RandomForest, ma RandomForest è più facile da implementare. In RandomForest, l’unico parametro di regolazione è il numero di alberi; mentre nel boosting, sono richiesti più parametri di regolazione oltre al numero di alberi, inclusi il restringimento e la profondità di interazione.
Se volete saperne di più, assicuratevi di dare un’occhiata al nostro corso Machine Learning Toolbox per R.