Inleiding
Beslissingsbomen zijn een soort algoritmen voor begeleid leren die zowel bij regressie- als bij classificatieproblemen kunnen worden gebruikt. Het werkt voor zowel categorische als continue input- en outputvariabelen.
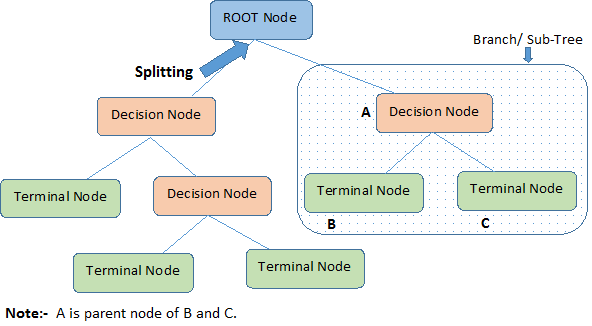
Laten we belangrijke terminologieën over Decision Tree identificeren, kijkend naar de bovenstaande afbeelding:
-
Root Node vertegenwoordigt de gehele populatie of steekproef. Deze wordt verder verdeeld in twee of meer homogene groepen.
-
Splitsing is een proces waarbij een knooppunt wordt verdeeld in twee of meer subknooppunten.
-
Wanneer een subknooppunt zich opsplitst in verdere subknooppunten, wordt dit een Beslissingsknooppunt genoemd.
-
Nodes die zich niet splitsen, worden een eindnode of blad genoemd.
-
Wanneer u sub-nodes van een beslissingsnode verwijdert, wordt dit proces snoeien genoemd. Het tegenovergestelde van snoeien is Splitsen.
-
Een deel van een hele boom wordt vertakking genoemd.
-
Een knooppunt dat in subknooppunten is verdeeld, wordt een moederknooppunt van de subknooppunten genoemd; terwijl de subknooppunten het kind van het moederknooppunt worden genoemd.
Typen beslissingsbomen
Regressiebomen
Laten we eens kijken naar de onderstaande afbeelding, die helpt bij het visualiseren van de aard van de partitionering die door een Regressieboom wordt uitgevoerd. Deze toont een niet-gesnoeide boom en een regressieboom die past op een willekeurige dataset. Beide visualisaties tonen een reeks splitsingsregels, beginnend bij de top van de boom. Merk op dat elke splitsing van het domein is uitgelijnd met een van de kenmerkassen. Het concept van asparallel splitsen veralgemeent zich rechttoe rechtaan naar dimensies groter dan twee. Voor een feature-ruimte met een grootte van $p$, een subset van $\mathbb{R}^p$, wordt de ruimte verdeeld in $M$ regio’s, $R_{m}$, die elk een $p$-dimensionaal “hyperblock” vormen.
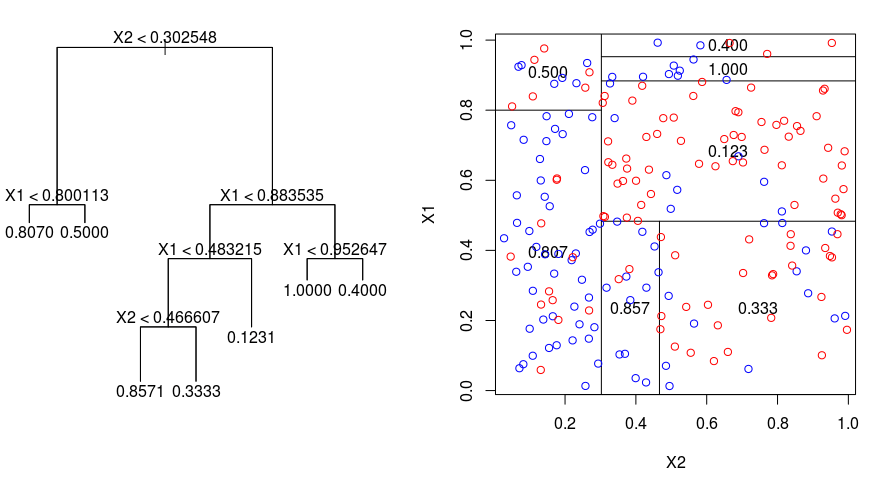
Om een regressieboom te bouwen, wordt eerst recursief binair splitsen gebruikt om een grote boom te laten groeien op de trainingsdata, waarbij pas wordt gestopt wanneer elk eindknooppunt minder dan een bepaald minimumaantal waarnemingen heeft. Recursief binair splitsen is een hebzuchtig en top-down algoritme dat gebruikt wordt om de residuele som van kwadraten (RSS) te minimaliseren, een foutmaat die ook gebruikt wordt in lineaire regressie-instellingen. De RSS, in het geval van een gepartitioneerde feature-ruimte met M partities, wordt gegeven door:
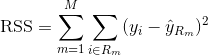
Beginnend bij de top van de boom, splitst u deze in 2 takken, waardoor een partitie van 2 ruimtes ontstaat. Vervolgens wordt deze splitsing aan de top van de boom meerdere malen uitgevoerd en wordt de splitsing van de kenmerken gekozen die de (huidige) RSS minimaliseert.
Naar aanleiding hiervan wordt de kostencomplexiteit op de grote boom gesnoeid om een reeks beste subtrees te verkrijgen, als functie van $³alpha$. Het basisidee hier is de invoering van een extra afstemparameter, aangeduid met $\alpha$, die de diepte van de boom in evenwicht brengt met de mate waarin hij bij de trainingsgegevens past.
U kunt K-voudige kruisvalidatie gebruiken om $\alpha$ te kiezen. Deze techniek bestaat eenvoudigweg uit het verdelen van de trainingswaarnemingen in K vouwen om de testfoutmarge van de subtrees te schatten. Het doel is de boom te kiezen die tot de laagste foutmarge leidt.
Classificatiebomen
Een classificatieboom lijkt veel op een regressieboom, behalve dat hij wordt gebruikt om een kwalitatieve respons te voorspellen in plaats van een kwantitatieve.
Werk eraan dat voor een regressieboom de voorspelde respons voor een waarneming wordt gegeven door de gemiddelde respons van de trainingswaarnemingen die tot hetzelfde eindknooppunt behoren. Bij een classificatieboom daarentegen voorspelt u dat elke waarneming behoort tot de meest voorkomende klasse van trainingswaarnemingen in de regio waartoe ze behoort.
Bij de interpretatie van de resultaten van een classificatieboom bent u vaak niet alleen geïnteresseerd in de klassevoorspelling die overeenkomt met een bepaalde eindknooppuntregio, maar ook in de klasseverhoudingen onder de trainingswaarnemingen die in die regio vallen.
De taak om een classificatieboom te laten groeien, lijkt sterk op de taak om een regressieboom te laten groeien. Net als bij de regressiemethode wordt een classificatieboom gekweekt met behulp van recursieve binaire splitsingen. In de classificatie-instelling kan de residuele som van kwadraten echter niet worden gebruikt als criterium voor het maken van de binaire splitsingen. In plaats daarvan kunt u een van de volgende drie methoden gebruiken:
- Classificatie-foutpercentage: In plaats van te kijken hoe ver een numerieke respons van de gemiddelde waarde afligt, zoals in de regressie-instelling, kunt u in plaats daarvan de “hit rate” definiëren als de fractie van de trainingswaarnemingen in een bepaald gebied die niet tot de meest voorkomende klasse behoren. De fout wordt gegeven door de volgende vergelijking:
E = 1 – argmaxc($\hat{\pi}_{mc}$)
waarin $\hat{\pi}_{mc}$ staat voor de fractie van de trainingsgegevens in regio Rm die tot klasse c behoren.
- Gini-index: De Gini-index is een alternatieve foutenmethode die aangeeft hoe “zuiver” een regio is. “Zuiverheid” betekent in dit geval hoeveel van de trainingsgegevens in een bepaalde regio tot één klasse behoren. Als een regio Rm gegevens bevat die grotendeels tot één klasse c behoren, zal de waarde van de Gini Index klein zijn:
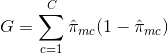
- Kruis-Entropie: Een derde alternatief, dat vergelijkbaar is met de Gini-index, staat bekend als de kruiselingse entropie of deviantie:
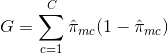
De kruiselingse entropie zal een waarde aannemen die dicht bij nul ligt als de ${{\pi}_{mc}$’s allemaal dicht bij 0 of dicht bij 1 liggen. Net als de Gini-index zal de kruiselingse entropie dus een kleine waarde aannemen als het m-de knooppunt zuiver is. In feite blijkt dat de Gini-index en de kruis-entropie numeriek vrij gelijkaardig zijn.
Bij het bouwen van een classificatieboom wordt gewoonlijk ofwel de Gini-index ofwel de kruis-entropie gebruikt om de kwaliteit van een bepaalde splitsing te evalueren, aangezien zij gevoeliger zijn voor de zuiverheid van knooppunten dan het classificatiefoutenpercentage. Elk van deze drie benaderingen kan worden gebruikt bij het snoeien van de boom, maar de classificatiefoutmarge verdient de voorkeur als de voorspellingsnauwkeurigheid van de uiteindelijke gesnoeide boom het doel is.
Voordelen en nadelen van beslissingsbomen
Het grote voordeel van beslissingsbomen is dat zij intuïtief zeer gemakkelijk uit te leggen zijn. In vergelijking met andere regressie- en classificatiebenaderingen sluiten zij nauw aan bij de menselijke besluitvorming. Ze kunnen grafisch worden weergegeven, en ze kunnen gemakkelijk omgaan met kwalitatieve voorspellers zonder de noodzaak om dummy variabelen te creëren.
Beslissingsbomen hebben over het algemeen echter niet hetzelfde niveau van voorspellende nauwkeurigheid als andere benaderingen, omdat ze niet helemaal robuust zijn. Een kleine verandering in de gegevens kan een grote verandering in de uiteindelijke geschatte boom veroorzaken.
Door veel beslisbomen samen te voegen, met behulp van methoden zoals bagging, random forests en boosting, kan de voorspellende prestatie van beslisbomen aanzienlijk worden verbeterd.
Boom-gebaseerde methoden
Bagging
De hierboven besproken beslisbomen hebben te lijden van een hoge variantie, wat betekent dat als je de trainingsdata willekeurig in tweeën deelt, en op beide helften een beslisboom toepast, de resultaten die je krijgt heel verschillend kunnen zijn. Een procedure met lage variantie daarentegen zal soortgelijke resultaten opleveren als ze herhaaldelijk op verschillende datasets wordt toegepast.
Bagging, of bootstrap-aggregatie, is een techniek die wordt gebruikt om de variantie van uw voorspellingen te verminderen door het resultaat van meerdere classifiers te combineren die op verschillende deelmonsters van dezelfde dataset zijn gemodelleerd. Hier is de vergelijking voor bagging:

waarbij u $B$ verschillende bootstrapped datasets genereert. Vervolgens train je je methode op de $bde$ bootstrapped trainingsset om $$bath{f}_{b}(x)$ te krijgen, en tenslotte het gemiddelde van de voorspellingen.
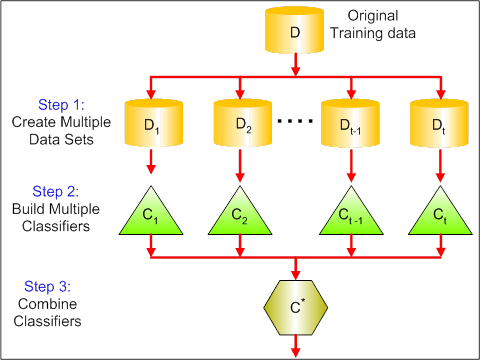
De onderstaande afbeelding toont de 3 verschillende stappen in bagging:
-
Stap 1: Hierbij vervang je de oorspronkelijke gegevens door nieuwe gegevens. De nieuwe gegevens bevatten gewoonlijk een fractie van de kolommen en rijen van de oorspronkelijke gegevens, die vervolgens kunnen worden gebruikt als hyperparameters in het baggingmodel.
-
Stap 2: U bouwt classifiers op elke dataset. Over het algemeen kunt u dezelfde classificator gebruiken voor het maken van modellen en voorspellingen.
-
Stap 3: Ten slotte gebruikt u een gemiddelde waarde om de voorspellingen van alle classificatoren te combineren, afhankelijk van het probleem. Over het algemeen zijn deze gecombineerde waarden robuuster dan een enkel model.
Hoewel bagging de voorspellingen voor veel regressie- en classificatiemethoden kan verbeteren, is het vooral nuttig voor beslisbomen. Om bagging toe te passen op regressie- en classificatiebomen worden $B$ regressie- en classificatiebomen gemaakt met behulp van $B$ bootstrapped trainingssets en wordt het gemiddelde van de voorspellingen berekend. Deze bomen worden diep gegroeid, en worden niet gesnoeid. Elke individuele boom heeft dus een hoge variantie, maar een lage bias. Het gemiddelde van deze $B$ bomen vermindert de variantie.
In het algemeen is aangetoond dat bagging een indrukwekkende verbetering van de nauwkeurigheid oplevert door honderden of zelfs duizenden bomen in één procedure te combineren.
Random Forests
Random Forests is een veelzijdige methode voor machinaal leren die zowel regressie- als classificatietaken kan uitvoeren. Het onderneemt ook dimensionale reductiemethoden, behandelt ontbrekende waarden, uitbijterwaarden en andere essentiële stappen van gegevensverkenning, en levert redelijk goed werk.
Random Forests biedt een verbetering ten opzichte van bagged trees door een kleine tweak die de bomen decorreleert. Net als bij bagging, bouw je een aantal beslisbomen op bootstrapped trainingsmonsters. Maar bij het bouwen van deze beslisbomen wordt telkens wanneer een splitsing in een boom wordt overwogen, een willekeurige steekproef van m voorspellers gekozen als splitsingskandidaten uit de volledige set van $p$ voorspellers. De splitsing mag slechts één van die $m$ voorspellers gebruiken. Dit is het belangrijkste verschil tussen random forests en bagging; want net als bij bagging is de keuze van de voorspeller $m = p$.
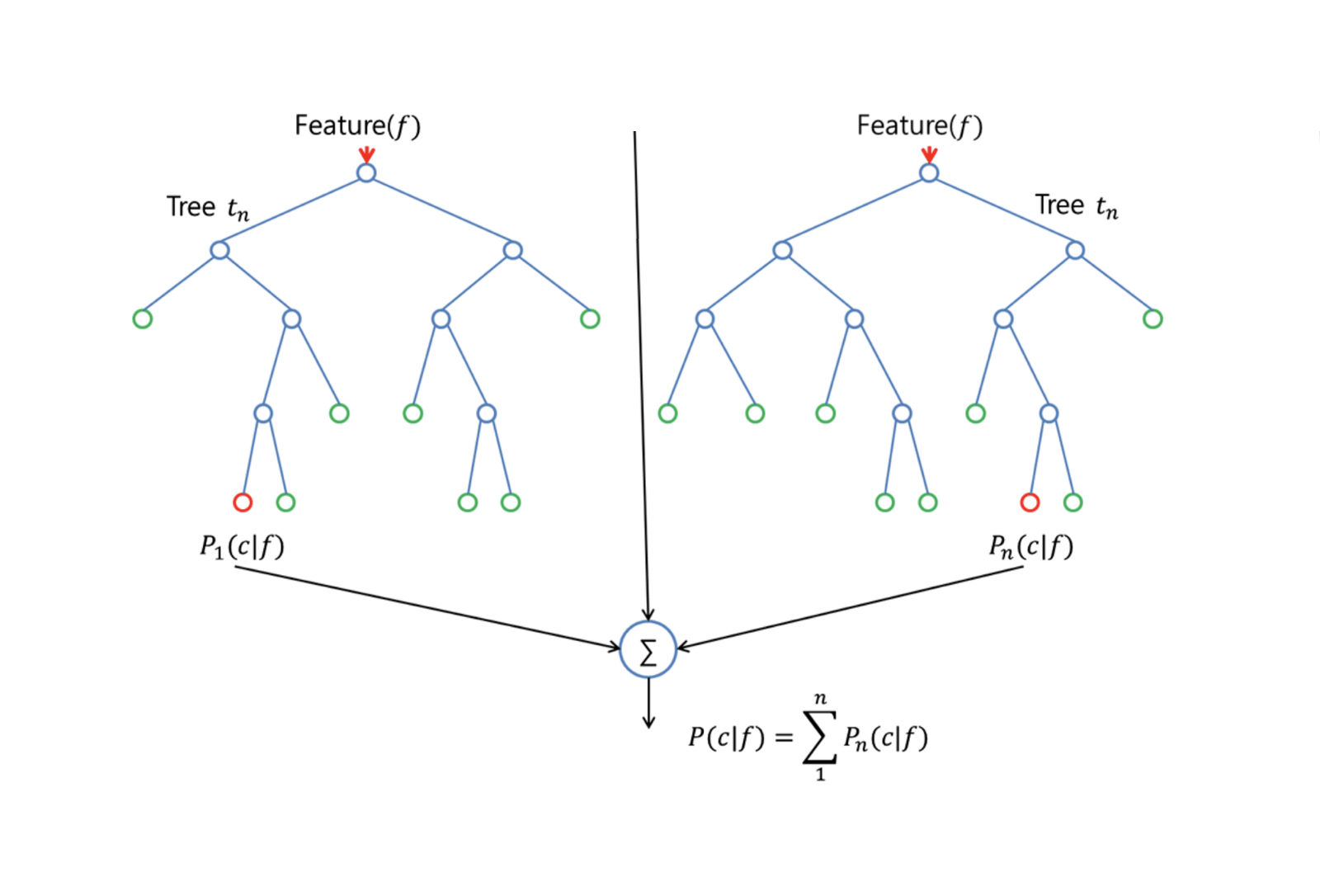
Om een random forest te kweken, moet u:
-
Eerst veronderstellen dat het aantal gevallen in de trainingsverzameling K is. Neem vervolgens een willekeurige steekproef van deze K gevallen en gebruik deze steekproef als de trainingsset om de boom te laten groeien.
-
Als er $p$ invoervariabelen zijn, geeft u een getal $m < p$ op zodat u bij elk knooppunt $m$ willekeurige variabelen uit de $p$ kunt selecteren. De beste splitsing op deze $m$ wordt gebruikt om de node te splitsen.
-
Elke boom wordt vervolgens zo groot mogelijk gegroeid en hoeft niet te worden gesnoeid.
-
Tot slot worden de voorspellingen van de doelbomen samengevoegd om nieuwe gegevens te voorspellen.
Random Forests is zeer effectief in het schatten van ontbrekende gegevens en het behouden van nauwkeurigheid wanneer een groot deel van de gegevens ontbreekt. Het kan ook fouten in evenwicht brengen in datasets waar de klassen onevenwichtig zijn. Het belangrijkste is dat het kan omgaan met massieve datasets met grote dimensionaliteit. Een nadeel van het gebruik van Random Forests is echter dat u gemakkelijk ruisende datasets kunt over-fitten, vooral in het geval van regressie.
Boosting
Boosting is een andere benadering om de voorspellingen die uit een beslisboom komen, te verbeteren. Net als bagging en random forests is het een algemene benadering die kan worden toegepast op vele statistische leermethoden voor regressie of classificatie. Bedenk dat bagging inhoudt dat meerdere kopieën van de oorspronkelijke trainingsdataset worden gemaakt met behulp van de bootstrap, dat op elke kopie een afzonderlijke beslissingsboom wordt gemonteerd en dat vervolgens alle bomen worden gecombineerd om één voorspellend model te maken. Met name wordt elke boom gebouwd op een bootstrap-dataset, onafhankelijk van de andere bomen.
Boosting werkt op een vergelijkbare manier, behalve dat de bomen sequentieel worden gekweekt: elke boom wordt gekweekt met behulp van informatie van eerder gekweekte bomen. Bij boosting wordt geen gebruik gemaakt van bootstrap sampling; in plaats daarvan wordt elke boom aangepast aan een gewijzigde versie van de oorspronkelijke dataset.

Voor zowel regressie- als classificatiebomen werkt boosting als volgt:
-
In tegenstelling tot het aanpassen van één grote beslissingsboom aan de gegevens, wat neerkomt op het hard aanpassen van de gegevens en mogelijk overfitting, leert de boosting-aanpak in plaats daarvan langzaam.
-
Gegeven het huidige model, past u een beslissingsboom aan op de residuen van het model. Dat wil zeggen, u past een boom met de huidige residuen, in plaats van de uitkomst $Y$, als respons.
-
U voegt vervolgens deze nieuwe beslisboom toe aan de passende functie om de residuen bij te werken. Elk van deze bomen kan vrij klein zijn, met slechts enkele eindknooppunten, die worden bepaald door de parameter $d$ in het algoritme. Door kleine bomen op de residuen in te passen, wordt $\hat{f}$ langzaam verbeterd in gebieden waar het niet goed presteert.
-
De krimpparameter $\nu$ vertraagt het proces nog verder, zodat meer en anders gevormde bomen de residuen kunnen aanvallen.
Boosting is erg nuttig wanneer je veel gegevens hebt en verwacht dat de beslisbomen erg complex zullen zijn. Boosting is gebruikt om veel uitdagende classificatie- en regressieproblemen op te lossen, waaronder risicoanalyse, sentimentanalyse, voorspellende reclame, prijsmodellering, verkoopschatting en patiëntendiagnose.
Beslissingsbomen in R
Classificatiebomen
Voor dit deel werk je met de Carseats-dataset met behulp van het tree-pakket in R. Denk eraan dat je de ISLR– en tree-pakketten eerst moet installeren in je R Studio-omgeving. Laten we eerst het Carseats dataframe laden uit het ISLR pakket.
library(ISLR)data(package="ISLR")carseats<-CarseatsLaten we ook het tree pakket laden.
require(tree)De Carseats-dataset is een dataframe met 400 waarnemingen over de volgende 11 variabelen:
-
Verkoop: verkoop per eenheid in duizenden
-
CompPrijs: prijs van de concurrent op elke locatie
-
Inkomen: inkomensniveau in de gemeenschap in 1000-en dollars
-
Advertenties: plaatselijk advertentiebudget op elke locatie in 1000-en dollars
-
Bevolking: regionale bevolking in duizenden
-
Prijs: prijs voor autostoeltjes op elke locatie
-
ShelveLoc: Slecht, Goed of Gemiddeld geeft de kwaliteit van de schaplocatie aan
-
Leeftijd: leeftijdsniveau van de bevolking
-
Opleiding: ed-niveau op de locatie
-
Urban: Ja/Nee
-
US: Ja/Nee
names(carseats)Laten we eens kijken naar het histogram van de autoverkoop:
hist(carseats$Sales)Opgemerkt moet worden dat Sales een kwantitatieve variabele is. U wilt dit aantonen met bomen met een binair antwoord. Daartoe maakt u van Sales een binaire variabele, die High wordt genoemd. Als de verkoop minder dan 8 is, zal het niet hoog zijn. Anders zal het hoog zijn. Dan kun je die nieuwe variabele High weer in het dataframe zetten.
High = ifelse(carseats$Sales<=8, "No", "Yes")carseats = data.frame(carseats, High)Nu gaan we een model vullen met behulp van beslisbomen. Natuurlijk kan je de variabele Sales hier niet hebben omdat je responsvariabele High werd gecreëerd uit Sales. Dus, laten we het uitsluiten en de boom passen.
tree.carseats = tree(High~.-Sales, data=carseats)Laten we de samenvatting van uw classificatieboom zien:
summary(tree.carseats)U kunt de betrokken variabelen zien, het aantal eindknooppunten, de residuele gemiddelde afwijking, evenals het misclassificatie foutpercentage. Om het visueler te maken, laten we de boom ook plotten, en dan annoteren met de handige text functie:
plot(tree.carseats)text(tree.carseats, pretty = 0)Er zijn zoveel variabelen, dat het erg ingewikkeld is om naar de boom te kijken. U kunt tenminste zien dat op elk van de eindknopen, ze zijn gelabeld Yes of No. Bij elk splitsingsknooppunt worden de variabelen en de waarde van de splitsingskeuze getoond (bijvoorbeeld Price < 92.5 of Advertising < 13.5).
Voor een gedetailleerde samenvatting van de boom kunt u deze gewoon afdrukken. Dat is handig als u details uit de boom wilt halen voor andere doeleinden:
tree.carseatsHet is tijd om de boom te snoeien. Laten we een training set en een test maken door het carseats dataframe op te splitsen in 250 training en 150 test samples. Eerst stel je een zaadje in om de resultaten reproduceerbaar te maken. Dan neem je een willekeurige steekproef van de ID (index) nummers van de steekproeven. Specifiek hier, je neemt een steekproef uit de set 1 tot n rij aantal rijen autostoelen, dat is 400. U wilt een steekproef van grootte 250 (standaard, steekproef gebruikt zonder vervanging).
set.seed(101)train=sample(1:nrow(carseats), 250)Dus nu krijgt u deze index van train, die 250 van de 400 waarnemingen indexeert. U kunt het model met tree reconstrueren, met gebruikmaking van dezelfde formule, behalve dat u de boom vertelt een deelverzameling te gebruiken die gelijk is aan train. Laten we dan een plot maken:
tree.carseats = tree(High~.-Sales, carseats, subset=train)plot(tree.carseats)text(tree.carseats, pretty=0)De plot ziet er een beetje anders uit vanwege de iets andere dataset. Niettemin ziet de complexiteit van de boom er ongeveer hetzelfde uit.
Nu ga je deze boom nemen en hem voorspellen op de testset, met behulp van de predict methode voor bomen. Hier wilt u de class labels daadwerkelijk voorspellen.
tree.pred = predict(tree.carseats, carseats, type="class")Daarna kunt u de fout evalueren met behulp van een misclassificatietabel.
with(carseats, table(tree.pred, High))Op de diagonalen staan de juiste classificaties, terwijl buiten de diagonalen de onjuiste staan. U wilt alleen de juiste classificaties herberekenen. Om dat te doen, kun je de som van de 2 diagonalen nemen, gedeeld door het totaal (150 testwaarnemingen).
(72 + 43) / 150Ok, je krijgt een fout van 0.76 met deze boom.
Wanneer je een grote bossige boom kweekt, zou hij te veel variantie kunnen hebben. Laten we dus kruisvalidatie gebruiken om de boom optimaal te snoeien. Met cv.tree gebruikt u de misclassificatiefout als basis voor het snoeien.
cv.carseats = cv.tree(tree.carseats, FUN = prune.misclass)cv.carseatsUittreksel van de resultaten toont de details van het pad van de kruisvalidatie. U ziet de grootte van de bomen als ze werden teruggesnoeid, de afwijkingen als het snoeien ging, evenals de kosten complexiteit parameter gebruikt in het proces.
Laten we dit uitzetten:
plot(cv.carseats)Kijkend naar de plot, zie je een neerwaartse spiraal deel als gevolg van de verkeerde classificatie fout op 250 kruisgevalideerde punten. Laten we dus een waarde kiezen in de neerwaartse stappen (12). Laten we vervolgens de boom snoeien tot een grootte van 12 om die boom te identificeren. Laten we tenslotte die boom plotten en annoteren om het resultaat te zien.
prune.carseats = prune.misclass(tree.carseats, best = 12)plot(prune.carseats)text(prune.carseats, pretty=0)Het is een beetje ondieper dan vorige bomen, en je kunt de labels echt lezen. Laten we het evalueren op de test dataset opnieuw.
tree.pred = predict(prune.carseats, carseats, type="class")with(carseats, table(tree.pred, High))(74 + 39) / 150Het lijkt erop dat de juiste classificaties een beetje gedaald. Het heeft ongeveer hetzelfde gedaan als uw oorspronkelijke boom, dus snoeien heeft niet veel kwaad gedaan met betrekking tot misclassificatie fouten, en gaf een eenvoudiger boom.
Vaak geven bomen geen erg goede voorspelfouten, dus laten we eens kijken naar random forests en boosting, die de neiging hebben beter te presteren dan bomen wat betreft voorspelling en misclassificatie.
Random Forests
Voor dit deel gaat u de Boston housing data gebruiken om random forests en boosting te onderzoeken. De dataset bevindt zich in het MASS-pakket. Het geeft de woningwaarden en andere statistieken in elk van de 506 buitenwijken van Boston op basis van een volkstelling in 1970.
library(MASS)data(package="MASS")boston<-Bostondim(boston)names(boston)Laten we ook het randomForest pakket laden.
require(randomForest)Om gegevens voor te bereiden voor random forest, laten we de seed instellen en een voorbeeld trainingsset van 300 observaties maken.
set.seed(101)train = sample(1:nrow(boston), 300)In deze dataset zijn er 506 buitenwijken van Boston. Voor elke wijk zijn er variabelen zoals criminaliteit per hoofd van de bevolking, soorten industrie, gemiddeld aantal kamers per woning, gemiddeld aandeel van de leeftijd van de huizen, enz. Laten we medv – de mediaanwaarde van door de eigenaar bewoonde woningen voor elk van deze buitenwijken, als de responsvariabele gebruiken.
Laten we een willekeurig bos passen en kijken hoe goed het presteert. Zoals gezegd, je gebruikt de respons medv, de mediane woningwaarde (in $1K dollars), en de training sample set.
rf.boston = randomForest(medv~., data = boston, subset = train)rf.bostonUitprinten van het random forest geeft de samenvatting: het aantal bomen (500 werden gekweekt), de gemiddelde gekwadrateerde residuen (MSR), en het percentage van de variantie verklaard. De MSR en % verklaarde variantie zijn gebaseerd op de out-of-bag schattingen, een heel slim apparaat in random forests om eerlijke fout schattingen te krijgen.
De enige tuning parameter in een random Forests is het argument genaamd mtry, dat is het aantal variabelen dat wordt geselecteerd bij elke splitsing van elke boom wanneer je een splitsing maakt. Zoals hier te zien is, is mtry 4 van de 13 verkennende variabelen (medv niet meegerekend) in de gegevens van Boston Housing – wat betekent dat telkens wanneer de boom een knooppunt komt splitsen, 4 variabelen willekeurig worden geselecteerd, waarna de splitsing wordt beperkt tot 1 van die 4 variabelen. Dat is hoe randomForests de bomen de-correleert.
Je gaat een serie willekeurige bossen fitten. Er zijn 13 variabelen, dus laten we mtry variëren van 1 tot 13:
-
Om de fouten vast te leggen, stel je 2 variabelen
oob.errentest.errop. -
In een lus van
mtryvan 1 tot 13, pas je eerst derandomForestmet die waarde vanmtryop detraindataset, waarbij je het aantal bomen beperkt tot 350. -
Vervolgens extraheert u de gemiddelde-kwadraatfout op het object (de out-of-bag-fout).
-
Vervolgens voorspelt u op de testdataset (
boston) met behulp vanfit(de fit vanrandomForest). -
Tot slot berekent u de testfout: de gemiddelde kwadratische fout, die gelijk is aan
mean( (medv - pred) ^ 2 ).
oob.err = double(13)test.err = double(13)for(mtry in 1:13){ fit = randomForest(medv~., data = boston, subset=train, mtry=mtry, ntree = 350) oob.err = fit$mse pred = predict(fit, boston) test.err = with(boston, mean( (medv-pred)^2 ))}Basaal gezien hebt u zojuist 4550 bomen gekweekt (13 keer 350). Laten we nu een plot maken met het matplot commando. De testfout en de out-of-bag fout worden samengebonden om een 2-koloms matrix te maken. Er zijn nog een paar andere argumenten in de matrix, waaronder de plot-tekenwaarden (pch = 23 betekent gevulde ruit), kleuren (rood en blauw), type gelijk aan beide (beide punten plotten en ze verbinden met de lijnen), en naam van de y-as (Mean Squared Error). U kunt ook een legende plaatsen in de rechter bovenhoek van de plot.
matplot(1:mtry, cbind(test.err, oob.err), pch = 23, col = c("red", "blue"), type = "b", ylab="Mean Squared Error")legend("topright", legend = c("OOB", "Test"), pch = 23, col = c("red", "blue"))Op zich zouden deze 2 curven met elkaar in overeenstemming moeten zijn, maar het lijkt erop dat de testfout een beetje lager is. Er zit echter veel variatie in deze testfoutenschattingen. Aangezien de out-of-bag foutenschatting is berekend op één dataset en de testfoutenschatting is berekend op een andere dataset, vallen deze verschillen vrij goed binnen de standaardfouten.
Merkt u dat de rode curve vloeiend boven de blauwe curve ligt? Deze foutenschattingen zijn zeer gecorreleerd, want de randomForest met mtry = 4 lijkt sterk op die met mtry = 5. Daarom is elk van de curven vrij vloeiend. Wat je ziet is dat mtry rond 4 de meest optimale keuze lijkt te zijn, althans voor de testfout. Deze waarde van mtry voor de out-of-bag fout is gelijk aan 9.
Dus met heel weinig tiers heb je een zeer krachtig voorspellingsmodel met random forests gemaakt. Hoe dat zo? De linkerzijde toont de prestatie van een enkele boom. De gemiddelde gekwadrateerde fout op out-of-bag is 26, en je hebt het teruggebracht tot ongeveer 15 (net iets boven de helft). Dit betekent dat je de fout met de helft hebt teruggebracht. Hetzelfde geldt voor de testfout, die is teruggebracht van 20 naar 12.
Boosting
Vergeleken met random forests, kweekt boosting kleinere en stuggere bomen en gaat op de bias af. Je gebruikt het pakket GBM (Gradient Boosted Modeling), in R.
require(gbm)GBM vraagt om de verdeling, die Gaussisch is, omdat je kwadratische fout verlies gaat doen. Je gaat GBM vragen om 10.000 bomen, wat klinkt als veel, maar dit zullen ondiepe bomen zijn. Interactie diepte is het aantal splitsingen, dus je wilt 4 splitsingen in elke boom. Shrinkage is 0.01, dat is hoeveel je de boom stap terug gaat verkleinen.
boost.boston = gbm(medv~., data = boston, distribution = "gaussian", n.trees = 10000, shrinkage = 0.01, interaction.depth = 4)summary(boost.boston)De summary functie geeft een variabele belangrijkheid plot. Het lijkt erop dat er 2 variabelen zijn die een hoog relatief belang hebben: rm (aantal kamers) en lstat (percentage mensen met een lagere economische status in de gemeenschap). Laten we deze 2 variabelen eens uitzetten:
plot(boost.boston,i="lstat")plot(boost.boston,i="rm")De 1e plot laat zien dat hoe hoger het percentage mensen met een lagere status in de voorstad, hoe lager de waarde van de huizenprijzen. De 2e plot toont de omgekeerde relatie met het aantal kamers: het gemiddelde aantal kamers in het huis neemt toe naarmate de prijs stijgt.
Het is tijd om een boosted model te voorspellen op de test dataset. Laten we eens kijken naar de testprestaties als functie van het aantal bomen:
-
Eerst maak je een raster van het aantal bomen in stappen van 100 van 100 tot 10.000.
-
Daarna voer je de functie
predictuit op het boosted model. Deze functie neemtn.treesals argument, en produceert een matrix van voorspellingen op de testgegevens. -
De afmetingen van de matrix zijn 206 testwaarnemingen en 100 verschillende voorspellingsvectoren bij de 100 verschillende waarden van boom.
n.trees = seq(from = 100, to = 10000, by = 100)predmat = predict(boost.boston, newdata = boston, n.trees = n.trees)dim(predmat)
Het is tijd om de testfout te berekenen voor elk van de voorspellingsvectoren:
-
predmatis een matrix,medvis een vector, dus (predmat–medv) is een matrix van verschillen. U kunt de functieapplygebruiken om de kolommen van deze kwadratische verschillen (het gemiddelde) te berekenen. Dat zou de kolomgewijze gemiddelde kwadratische fout voor de voorspellingsvectoren berekenen. -
Dan maakt u een plot met soortgelijke parameters als die welke voor Random Forest worden gebruikt. Het zou een boosting error plot laten zien.
boost.err = with(boston, apply( (predmat - medv)^2, 2, mean) )plot(n.trees, boost.err, pch = 23, ylab = "Mean Squared Error", xlab = "# Trees", main = "Boosting Test Error")abline(h = min(test.err), col = "red")
De boosting error daalt vrij sterk als het aantal bomen toeneemt. Dit is een bewijs dat boosting niet geneigd is tot overfit. Laten we ook de beste testfout van het randomForest in de plot opnemen. Boosting komt in feite een redelijk eind onder de testfout van randomForest.
Conclusie
Dus dat is het einde van deze R tutorial over het bouwen van beslisboommodellen: classificatiebomen, random forests, en boosted trees. De laatste 2 zijn krachtige methoden die je altijd kunt gebruiken als dat nodig is. In mijn ervaring presteert boosting meestal beter dan RandomForest, maar RandomForest is gemakkelijker te implementeren. Bij RandomForest is de enige afstemmingsparameter het aantal bomen; terwijl bij boosting naast het aantal bomen meer afstemmingsparameters nodig zijn, waaronder de krimp en de interactiediepte.
Wilt u meer weten, kijk dan eens naar onze cursus Machine Learning Toolbox voor R.