DNA
DNA er en polymer, der består af monomerer kaldet deoxynucleotider. Monomeren indeholder et simpelt sukker (deoxyribose, vist med sort nedenfor), en fosfatgruppe (med rødt) og en cyklisk organisk R-gruppe (med blåt), der svarer til sidekæden i en aminosyre.
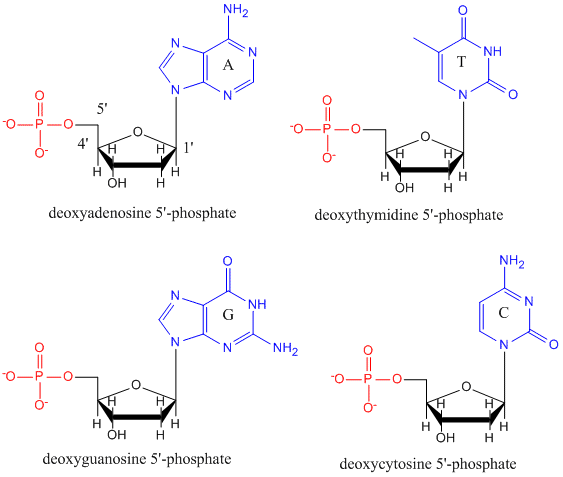
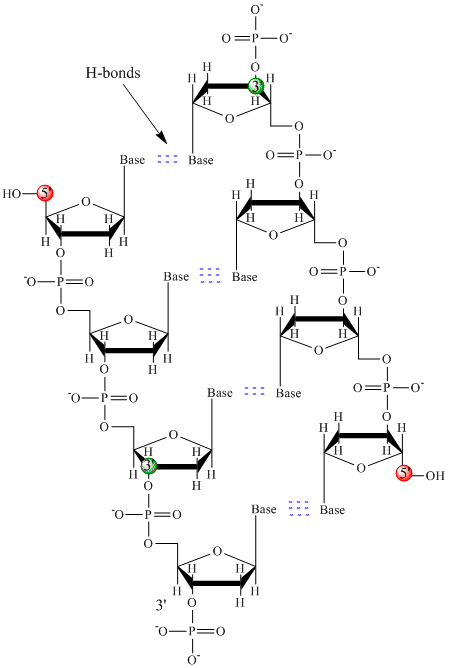
Der er kun fire baser i DNA (i modsætning til de 20 forskellige sidekæder i proteiner), som vi for enkelhedens skyld forkorter med A, G, C og T. De er baser, da de indeholder aminogrupper, der kan modtage protoner. Polymeren består af en sukker – fosfat – sukker – fosfat-rygsøjle med en base knyttet til hvert sukkermolekyle. Ligesom for proteiner er DNA-ryggen polær, men også ladet. Det er en polyanion. Baserne, der svarer til sidekæderne i aminosyrer, er overvejende polære. I betragtning af rygsøjlens ladede natur kan man forvente, at DNA ikke foldes til en kompakt kugleform (sfærisk), selv om positivt ladede kationer som Mg binder sig til og stabiliserer ladningen på polymeren. I stedet eksisterer DNA normalt som en dobbeltstrenget (ds) struktur, hvor sukker-fosfatryggen i de to forskellige strenge løber i modsatte retninger (5′-3′ og den anden 3′-5′). Strengene holdes sammen af hydrogenbindinger mellem baser på komplementære strenge. Ligesom proteiner har DNA således sekundær struktur, men i dette tilfælde er hydrogenbindingerne ikke inden for rygraden, men mellem “sidekæden”-baserne på de modsatte strenge. Det er faktisk en misvisende betegnelse at kalde dsDNA for et molekyle, da det i virkeligheden består af to forskellige, komplementære strenge, der holdes sammen af hydrogenbindinger. En struktur af ds-DNA, der viser strengenes modsatte polaritet, er vist nedenfor.
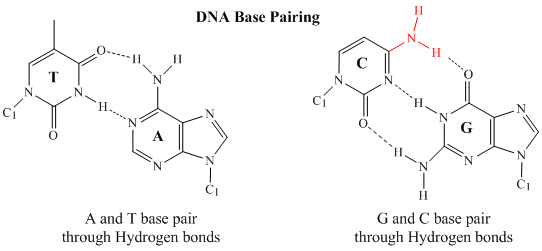
I dobbeltstrenget DNA kan guaninbasen (G) på den ene streng danne tre H-bindinger med en cytosinbase (C) på den anden streng (dette kaldes et GC-basepar). Thyminbasen (T) på den ene streng kan danne to H-bindinger med en adeninbase (A) på den anden streng (dette kaldes et AT-basepar). Dobbeltstrenget DNA har en regelmæssig geometrisk struktur med en fast afstand mellem de to backbones. Dette kræver, at baseparrene består af en base med en to-ring (bicyklisk) struktur (disse baser kaldes puriner) og en base med en enkelt ringstruktur (disse baser kaldes pyrimidiner). Derfor er et G og A eller et T og C ikke mulige baseparpartnere.
Dobbeltstrenget DNA varierer i længde (antal sukker-fosfat-enheder, der er forbundet), basesammensætning (hvor mange af hvert sæt baser) og sekvens (rækkefølgen af baserne i rygsøjlen). De følgende links indeholder interaktive Jmol-modeller af dsDNA fremstillet af Angel Herráez, Univ. de Alcalá (Spanien) og Eric Martz.
- Jmol-model af ds-DNA med basepar og H-bindinger
- Jmol-model af DNA-strenge og spiralformet rygsøjle
- Jmol-model af DNA-ender og parallelismer
Chromosomer består af ét dsDNA med mange forskellige bundne proteiner. Det menneskelige genom har ca. 3 milliarder basepar af dna. Derfor har hvert enkelt kromosom i et par i gennemsnit ca. 150 millioner basepar og masser af proteiner bundet til sig. dsDNA er et stærkt ladet molekyle og kan i første omgang betragtes som et langt stavlignende molekyle med en stor negativ ladning. Det er en polyanion. Dette meget store molekyle skal på en eller anden måde pakkes ind i en lille kerne i en lillebitte celle. I komplekse (eukaryote) celler løses dette pakningsproblem ved at vikle DNA rundt om et kernekompleks af fire forskellige par (i alt otte proteiner) af histonproteiner (H2A, H2B, H3 og H4), som har positive nettoladninger. Histon-kernekomplekset med dsDNA viklet omkring ca. 2,5 gange kaldes nukleosomet.
Jmol-model af nukleosomet
DNA kan antage to andre typer af dobbelt-helikale former. Den, der blev opdaget af Watson og Crick og findes i de fleste lærebøger, kaldes B-DNA. Afhængigt af den aktuelle DNA-sekvens og DNA’s hydreringstilstand kan det overtales til at danne to andre typer dobbeltstrenge-helixer, Z- og A-DNA. A-formen er meget mere åben end B-formen.
De 3,2 milliarder basepar af DNA i mennesker indeholder omkring 24.000 korte strækninger (gener), der koder for forskellige proteiner. Disse gener er spredt ud blandt DNA, som er med til at bestemme, om genet afkodes til RNA-molekyler (se nedenfor) og i sidste ende til proteiner. For at et bestemt gen kan aktiveres (eller “tændes”), skal specifikke proteiner binde sig til området i et bestemt gen. Hvordan kan bindingsproteiner finde specifikke bindingsmål blandt det enorme antal basepar, der tilnærmelsesvis har en gentagelse af en gentagelse af sukker-fosfat-base-basen? Jmol nedenfor viser, hvordan man kan opnå specificitet. Når DNA vikler sig ind i en dobbeltspiral gennem basepar mellem AT og GC, er der stadig hydrogenbindingsdonorer (amid Hs) og -acceptorer (Os) på de baser, der ikke anvendes i intrastrand baseparring, til rådighed i ds-DNA-helixens major og minor lunge (se Jmol nedenfor). Unikke baseparsekvenser vil vise unikke mønstre af H-båndsdonorer og -acceptorer i major grove. Disse donorer/acceptorer kan genkendes af specifikke DNA-bindingsproteiner, som ved binding kan føre til genaktivering.
- Jmol-model af dsDNA, der viser unikke H-båndsdonorer og -acceptorer i den store grove