El ADN
El ADN es un polímero, formado por monómeros llamados desoxinucleótidos. El monómero contiene un azúcar simple (desoxirribosa, mostrada en negro a continuación), un grupo fosfato (en rojo) y un grupo R orgánico cíclico (en azul) que es análogo a la cadena lateral de un aminoácido.
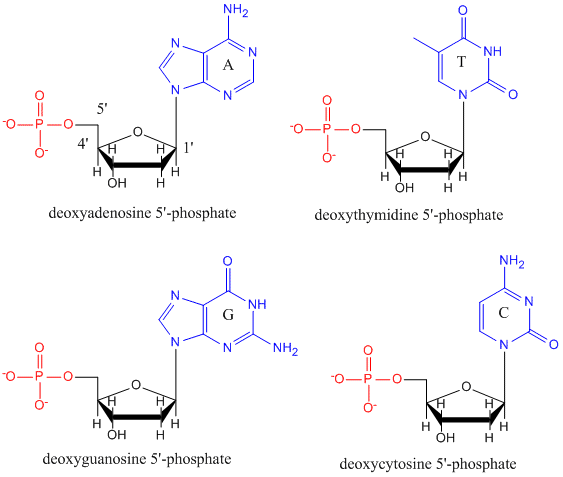
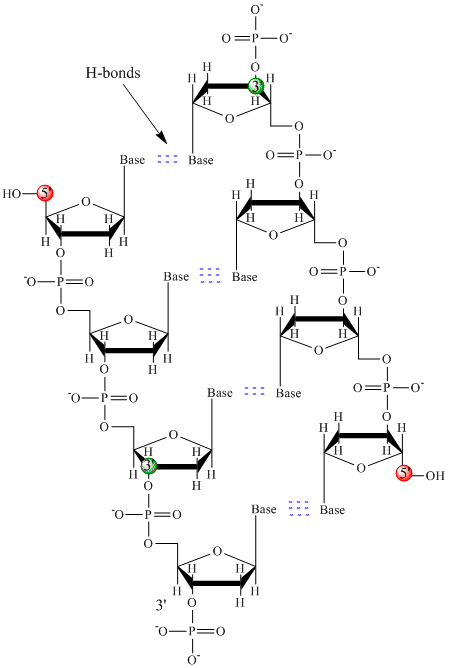
En el ADN sólo se utilizan cuatro bases (en contraste con las 20 cadenas laterales diferentes de las proteínas) que abreviaremos, para simplificar, como A, G, C y T. Son bases ya que contienen grupos aminos que pueden aceptar protones. El polímero consiste en un esqueleto de azúcar – fosfato – azúcar – fosfato, con una base unida a cada molécula de azúcar. Al igual que las proteínas, la columna vertebral del ADN es polar pero también está cargada. Es un polianión. Las bases, análogas a las cadenas laterales de los aminoácidos, son predominantemente polares. Dada la naturaleza cargada de la espina dorsal, es de esperar que el ADN no se pliegue a una forma globular (esférica) compacta, aunque los cationes cargados positivamente, como el Mg, se unan y estabilicen la carga del polímero. En su lugar, el ADN existe normalmente como una estructura de doble cadena (ds) con las columnas vertebrales de azúcar y fosfato de las dos cadenas diferentes que van en direcciones opuestas (5′-3′ y la otra 3′-5′). Las hebras se mantienen unidas por enlaces de hidrógeno entre las bases de las hebras complementarias. Por lo tanto, al igual que las proteínas, el ADN tiene una estructura secundaria, pero en este caso, los enlaces de hidrógeno no están dentro de la columna vertebral, sino entre las bases de la «cadena lateral» de las cadenas opuestas. En realidad es un error llamar al ADN-ds una molécula, ya que en realidad consiste en dos hebras diferentes y complementarias unidas por enlaces de hidrógeno. A continuación se muestra una estructura de ADN-ds que muestra la polaridad opuesta de las cadenas.
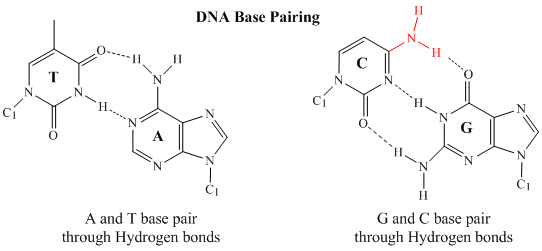
En el ADN de doble cadena, la base de guanina (G) de una cadena puede formar tres enlaces H con una base de citosina (C) de otra cadena (esto se llama un par de bases GC). La base timina (T) de una hebra puede formar dos enlaces H con una base adenina (A) de la otra hebra (lo que se denomina un par de bases AT). El ADN de doble cadena tiene una estructura geométrica regular con una distancia fija entre las dos cadenas. Esto requiere que los pares de bases estén formados por una base con una estructura de dos anillos (bicíclica) (estas bases se llaman purinas) y otra con una estructura de un solo anillo (estas bases se llaman pirimidinas). Por lo tanto, una G y una A o una T y una C no son posibles parejas de bases.
El ADN de doble cadena varía en longitud (número de unidades de azúcar-fosfato conectadas), composición de bases (cuántas de cada conjunto de bases) y secuencia (el orden de las bases en la columna vertebral). Los siguientes enlaces proporcionan modelos Jmol interactivos de dsDNA realizados por Angel Herráez, Univ. de Alcalá (España) y Eric Martz.
- Modelo Jmol de ADNs con pares de bases y enlaces H
- Modelo Jmol de hebras de ADN y columna vertebral helicoidal
- Modelo Jmol de extremos de ADN y paralelismos
Los cromosomas consisten en un ADNs con muchas proteínas diferentes unidas. El genoma humano tiene unos 3.000 millones de pares de bases de ADN. Por lo tanto, en promedio, cada uno de los cromosomas de un par tiene unos 150 millones de pares de bases y muchas proteínas unidas a él. El dsADN es una molécula altamente cargada, y puede verse, en una primera aproximación, como una molécula larga en forma de varilla con una gran carga negativa. Es un polianión. Esta molécula tan grande debe empaquetarse de algún modo en el pequeño núcleo de una célula diminuta. En las células complejas (eucariotas), este problema de empaquetamiento se resuelve enrollando el ADN alrededor de un complejo central de cuatro pares diferentes (ocho proteínas en total) de proteínas histónicas (H2A, H2B, H3 y H4) que tienen cargas positivas netas. El complejo del núcleo de histonas con el dsADN enrollado aproximadamente 2,5 veces se denomina nucleosoma.
Modelo Jmol del nucleosoma
El ADN puede adoptar otros dos tipos de formas doblemente helicoidales. La descubierta por Watson y Crick y que aparece en la mayoría de los libros de texto se denomina ADN-B. Dependiendo de la secuencia real del ADN y del estado de hidratación del mismo, puede ser obligado a formar otros dos tipos de hélices de doble cadena, el ADN Z y el A. La forma A es mucho más abierta que la forma B.
Los 3.200 millones de pares de bases de ADN de los seres humanos contienen unos 24.000 tramos cortos (genes) que codifican diferentes proteínas. Estos genes están intercalados en el ADN, lo que ayuda a determinar si el gen se descodifica en moléculas de ARN (véase más adelante) y, finalmente, en proteínas. Para que un gen concreto se active (o se «encienda»), las proteínas específicas deben unirse a la región de un gen concreto. ¿Cómo pueden las proteínas de unión encontrar objetivos de unión específicos entre el gran número de pares de bases que, en una primera aproximación, tienen una repetición de azúcar-fosfato-base? El Jmol de abajo muestra cómo se puede lograr la especificidad. Cuando el ADN se enrolla en una doble hélice a través de pares de bases entre AT y GC, los donantes de enlaces de hidrógeno (amida Hs) y los aceptores (Os) en las bases que no se utilizan en el emparejamiento de bases intracadena, siguen estando disponibles en el surco mayor y menor de la hélice de ADN-ds (véase el Jmol de abajo). Las secuencias de pares de bases únicas mostrarán patrones únicos de donantes y aceptores de enlaces H en el surco mayor. Estos donantes/aceptores pueden ser reconocidos por proteínas específicas de unión al ADN que, al unirse, pueden conducir a la activación del gen.
- Modelo Jmol de dsADN que muestra donantes y aceptores de enlaces H únicos en el surco mayor