DNA
DNA on polymeeri, joka koostuu monomeereistä, joita kutsutaan deoksinukleotideiksi. Monomeeri sisältää yksinkertaisen sokerin (deoksiriboosi, esitetty alla mustalla), fosfaattiryhmän (punaisella) ja syklisen orgaanisen R-ryhmän (sinisellä), joka on analoginen aminohapon sivuketjun kanssa.
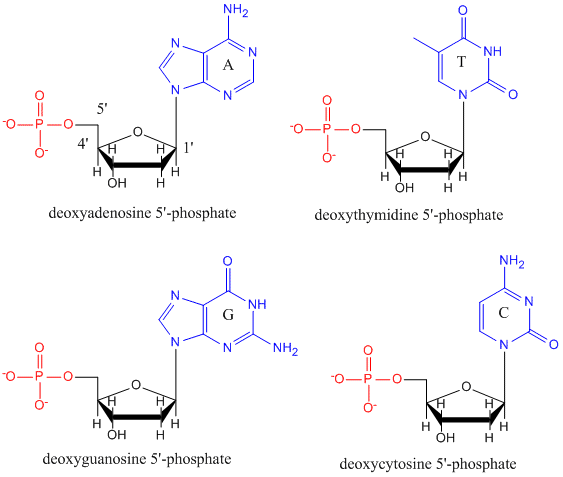
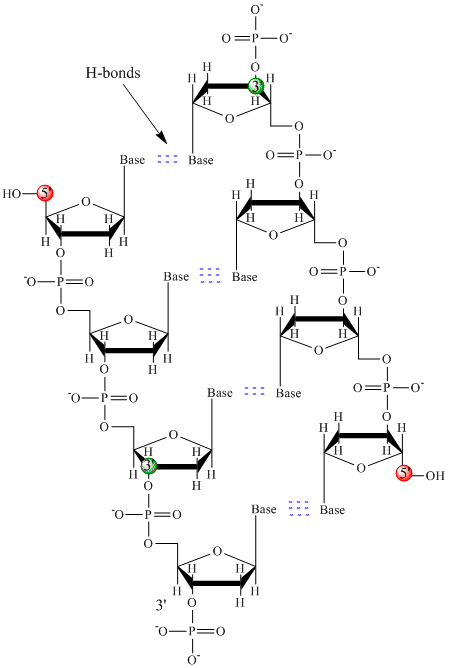
DNA:ssa käytetään vain neljää emästä (toisin kuin proteiineissa on 20 erilaista sivuketjua), jotka lyhennämme yksinkertaisuuden vuoksi nimillä A, G, C ja T. Ne ovat emäksiä, koska ne sisältävät amiiniryhmiä, jotka voivat ottaa vastaan protoneja. Polymeeri koostuu sokeri – fosfaatti – sokeri – fosfaattirungosta, jossa kuhunkin sokerimolekyyliin on kiinnittynyt yksi emäs. Kuten proteiineilla, DNA:n selkäranka on polaarinen mutta myös varautunut. Se on polyanioni. Emäkset, jotka ovat analogisia aminohappojen sivuketjujen kanssa, ovat pääasiassa poolisia. Selkärangan varautuneen luonteen vuoksi voisi olettaa, että DNA ei taitu tiiviiksi pallomaiseksi (pallomaiseksi) muodoksi, vaikka positiivisesti varautuneet kationit, kuten Mg, sitoutuvat polymeeriin ja stabiloivat sen varauksen. Sen sijaan DNA on yleensä kaksisäikeinen (ds) rakenne, jossa kahden eri säikeen sokeri-fosfaattirungot kulkevat vastakkaisiin suuntiin (5′-3′ ja toinen 3′-5′). Säikeet pysyvät yhdessä komplementaaristen säikeiden emästen välisten vetysidosten avulla. DNA:lla on siis proteiinien tavoin sekundaarirakenne, mutta tässä tapauksessa vetysidokset eivät ole selkärangan sisällä vaan vastakkaisten säikeiden ”sivuketjujen” emästen välillä. On itse asiassa väärin kutsua dsDNA:ta molekyyliksi, koska se koostuu kahdesta eri, toisiaan täydentävästä säikeestä, joita vetysidokset pitävät yhdessä. Alla on esitetty ds-DNA:n rakenne, jossa näkyy säikeiden vastakkainen napaisuus.
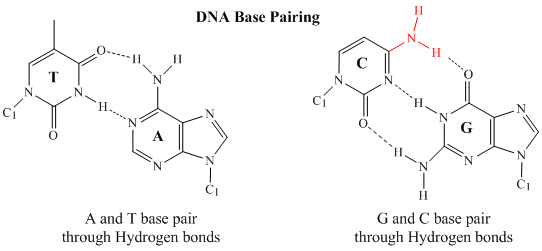
Kaksisäikeisessä DNA:ssa yhden säikeen guaniini- (G) emäs voi muodostaa kolme H-sidosta toisen säikeen sytosiini- (C) emäksen kanssa (tätä kutsutaan GC-emäspaariksi). Toisen säikeen tymiini-emäs (T) voi muodostaa kaksi H-sidosta toisen säikeen adeniini-emäksen (A) kanssa (tätä kutsutaan AT-emäspariksi). Kaksijuosteisella DNA:lla on säännöllinen geometrinen rakenne, jossa kahden selkärangan välillä on kiinteä etäisyys. Tämä edellyttää, että emäsparien on koostuttava yhdestä emäksestä, jolla on kaksirengasrakenne (näitä emäksiä kutsutaan puriineiksi), ja yhdestä emäksestä, jolla on yksirengasrakenne (näitä emäksiä kutsutaan pyrimidiiniksi). Näin ollen G ja A tai T ja C eivät ole mahdollisia emäsparien kumppaneita.
Kaksisäikeinen DNA vaihtelee pituuden (toisiinsa liittyvien sokeri-fosfaattiyksiköiden määrä), emäskoostumuksen (kuinka monta kutakin emäsryhmää on) ja järjestyksen (emästen järjestys selkärangassa) suhteen. Seuraavista linkeistä löytyvät Angel Herráezin, Univ. de Alcalá (Espanja) ja Eric Martzin tekemät interaktiiviset Jmol-mallit dsDNA:sta.
- Jmol-malli ds-DNA:sta, jossa on emäspareja ja H-sidoksia
- Jmol-malli DNA-säikeistä ja spiraalisesta selkärangasta
- Jmol-malli DNA:n päistä ja rinnakkaislohkoista
Kromosomit koostuvat yhdestä dsDNA:sta ja siihen sitoutuneista monista eri proteiineista. Ihmisen genomissa on noin 3 miljardia emäsparia DNA:ta. Näin ollen kromosomiparin jokaisessa yksittäisessä kromosomissa on keskimäärin noin 150 miljoonaa emäsparia ja siihen on sitoutunut paljon proteiineja. dsDNA on voimakkaasti varautunut molekyyli, ja sitä voidaan pitää ensimmäisessä likimääräisessä tarkastelussa pitkänä sauvamaisena molekyylinä, jolla on suuri negatiivinen varaus. Se on polyanioni. Tämä erittäin suuri molekyyli on jotenkin pakattava pienen solun pieneen ytimeen. Monimutkaisissa (eukaryoottisissa) soluissa tämä pakkautumisongelma ratkaistaan kietomalla DNA neljän eri histoniproteiiniparin (H2A, H2B, H3 ja H4) muodostaman ydinkompleksin ympärille (yhteensä kahdeksan proteiinia), joilla on positiivinen nettovaraus. Histoniydinkompleksia, jonka ympärille dsDNA on kierretty noin 2,5 kertaa, kutsutaan nukleosomiksi.
Jmol-malli nukleosomista
DNA voi omaksua kahden muunlaisen kaksoiskelikaalisen muodon. Watsonin ja Crickin löytämää ja useimmissa oppikirjoissa esiintyvää kutsutaan B-DNA:ksi. Todellisesta DNA-sekvenssistä ja DNA:n hydrataatiotilasta riippuen se voidaan taivutella muodostamaan kaksi muuta tyyppiä kaksoiskierteisiä kierteitä, Z- ja A-DNA:ta. A-muoto on paljon avoimempi kuin B-muoto.
Ihmisen 3,2 miljardia emäsparia DNA:ta sisältää noin 24 000 lyhyttä jaksoa (geeniä), jotka koodaavat erilaisia proteiineja. Nämä geenit ovat DNA:n välissä, mikä auttaa määrittämään, puretaanko geeni RNA-molekyyleiksi (ks. jäljempänä) ja lopulta proteiineiksi. Jotta tietty geeni aktivoituu (tai ”käynnistyy”), tiettyjen proteiinien on sitouduttava tietyn geenin alueeseen. Miten sitoutuvat proteiinit voivat löytää erityiset sitoutumiskohteet valtavasta määrästä emäspareja, joissa on alustavasti toistuvia sokeri-fosfaatti-emäspareja? Alla oleva Jmol osoittaa, miten spesifisyys voidaan saavuttaa. Kun DNA kietoutuu kaksoiskierteeksi AT:n ja GC:n välisten emäsparien välityksellä, ds-DNA-kierteen isossa ja pienessä urassa on edelleen käytettävissä vetysidoksen luovuttajia (amidi Hs) ja akseptoreita (Os) emäksissä, joita ei käytetä juosteen sisäisessä emäsparinmuodostuksessa (ks. Jmol alla). Ainutlaatuisissa emäsparisekvensseissä on ainutlaatuisia H-sidoksen luovuttajien ja akseptoijien kuvioita suuressa urassa. Spesifiset DNA:ta sitovat proteiinit voivat tunnistaa nämä luovuttajat/akseptorit, jotka sitoutuessaan voivat johtaa geenin aktivoitumiseen.
- Jmol-malli dsDNA:sta, jossa näkyy uniikkeja H-sidoksen luovuttajia ja akseptoreita suuressa urassa