DNA
DNA is een polymeer, dat bestaat uit monomeren die deoxynucleotiden worden genoemd. Het monomeer bevat een enkelvoudige suiker (deoxyribose, hieronder in zwart weergegeven), een fosfaatgroep (in rood), en een cyclische organische R-groep (in blauw) die analoog is aan de zijketen van een aminozuur.
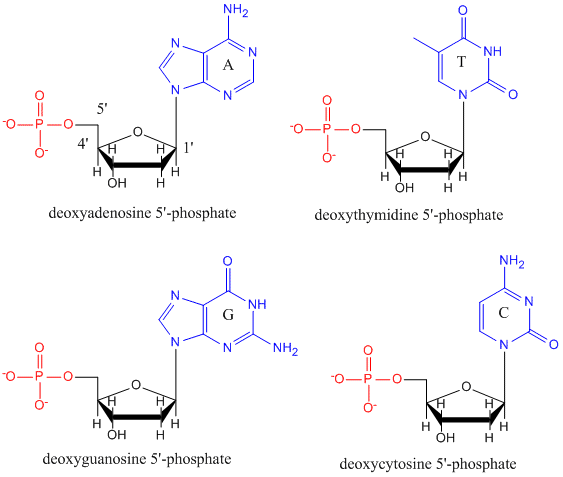
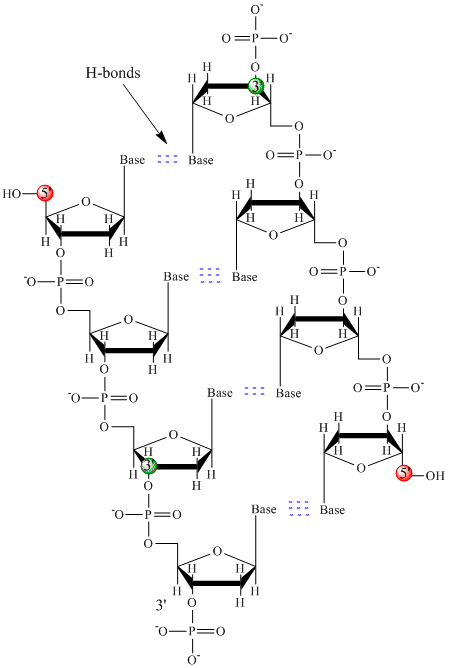
In DNA worden slechts vier basen gebruikt (in tegenstelling tot de 20 verschillende zijketens in eiwitten), die we voor het gemak zullen afkorten als A, G, C en T. Het zijn basen omdat ze aminegroepen bevatten die protonen kunnen accepteren. Het polymeer bestaat uit een suiker – fosfaat – suiker – fosfaat ruggengraat, met aan elk suikermolecuul een base. Net als bij eiwitten is de DNA-backbone polair, maar ook geladen. Het is een polyanion. De basen, naar analogie van de zijketens van aminozuren, zijn overwegend polair. Gezien de geladen aard van de ruggengraat zou je verwachten dat DNA zich niet tot een compacte bolvorm vouwt, ook al binden positief geladen kationen zoals Mg zich aan het polymeer en stabiliseren zij de lading op het polymeer. In plaats daarvan bestaat DNA meestal als een dubbelstrengs (ds) structuur, waarbij de suiker-fosfaat ruggengraten van de twee verschillende strengen in tegengestelde richtingen lopen (5′-3′ en de andere 3′-5′). De strengen worden bijeengehouden door waterstofbruggen tussen basen op complementaire strengen. Net als eiwitten heeft DNA dus een secundaire structuur, maar in dit geval bevinden de waterstofbruggen zich niet binnen de ruggengraat, maar tussen de basen van de “zijketens” op de tegenover elkaar liggende strengen. Het is eigenlijk een verkeerde benaming om ds-DNA een molecuul te noemen, omdat het in werkelijkheid bestaat uit twee verschillende, complementaire strengen die bij elkaar worden gehouden door waterstofbruggen. Een structuur van ds-DNA die de tegengestelde polariteit van de strengen laat zien, is hieronder afgebeeld.
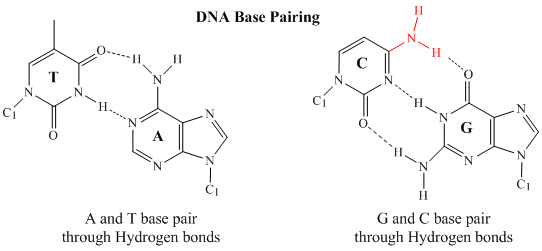
In dubbelstrengs DNA kan de guanine (G) base op de ene streng drie H-bindingen vormen met een cytosine (C) base op een andere streng (dit wordt een GC-basenpaar genoemd). De thymine (T) base op de ene streng kan twee H-bindingen vormen met een adenine (A) base op de andere streng (dit wordt een AT-basenpaar genoemd). Dubbelstrengs DNA heeft een regelmatige geometrische structuur met een vaste afstand tussen de twee ruggengraten. Dit betekent dat de basenparen moeten bestaan uit een base met een twee-rings (bicyclische) structuur (deze basen worden purines genoemd) en een base met een enkele ringstructuur (deze basen worden pyrimidines genoemd). Een G en een A of een T en een C zijn dus geen mogelijke basenpaarpartners.
Dubbelstrengs DNA varieert in lengte (aantal verbonden suiker-fosfaat eenheden), basensamenstelling (hoeveel van elke set basen) en sequentie (de volgorde van de basen in de ruggengraat). De volgende links geven interactieve Jmol modellen van dsDNA gemaakt door Angel Herráez, Univ. de Alcalá (Spanje) en Eric Martz.
- Jmol model van ds-DNA met basenparen en H-bindingen
- Jmol model van DNA strengen en spiraalvormige ruggengraat
- Jmol model van DNA uiteinden en parallellismen
Chromosomen bestaan uit één dsDNA met veel verschillende gebonden eiwitten. Het menselijk genoom heeft ongeveer 3 miljard basenparen DNA. Gemiddeld heeft elk chromosoom van een paar dus ongeveer 150 miljoen basenparen en heel veel eiwitten eraan gebonden. dsDNA is een sterk geladen molecuul, en kan bij wijze van eerste benadering worden gezien als een lang staafvormig molecuul met een grote negatieve lading. Het is een polyanion. Dit zeer grote molecuul moet op de een of andere manier in een kleine kern van een piepkleine cel worden ingepakt. In complexe (eukaryote) cellen wordt dit verpakkingsprobleem opgelost door DNA op te rollen rond een kerncomplex van vier verschillende paren (acht eiwitten in totaal) van histon-eiwitten (H2A, H2B, H3, en H4) die netto positieve ladingen hebben. Het histon-kerncomplex met dsDNA dat ongeveer 2,5 keer rond gewikkeld is, wordt het nucleosoom genoemd.
Jmol-model van het nucleosoom
DNA kan twee andere soorten dubbel-helicale vormen aannemen. De vorm die door Watson en Crick is ontdekt en in de meeste leerboeken voorkomt, wordt B-DNA genoemd. Afhankelijk van de werkelijke DNA-sequentie en de hydratatietoestand van het DNA, kan het zo worden gebracht dat het twee andere typen dubbelstrengs helices vormt, Z en A DNA. De A-vorm is veel opener dan de B-vorm.
De 3,2 miljard basenparen DNA in de mens bevatten ongeveer 24.000 korte stukken (genen) die coderen voor verschillende eiwitten. Deze genen zijn onderverdeeld in DNA dat mede bepaalt of het gen wordt gedecodeerd tot RNA-moleculen (zie hieronder) en uiteindelijk tot eiwitten. Om een bepaald gen te activeren (of “aan te zetten”), moeten specifieke eiwitten zich binden aan de regio van een bepaald gen. Hoe kunnen bindingsproteïnen specifieke bindingsdoelen vinden onder het enorme aantal basenparen die, bij een eerste benadering, een repetitieve herhaling van suiker-fosfaat-basen hebben? Onderstaande Jmol laat zien hoe specificiteit kan worden bereikt. Wanneer DNA zich tot een dubbele helix windt via basenparen tussen AT en GC, zijn er nog steeds waterstofbrug-donors (amide Hs) en -acceptors (Os) op de basen die niet gebruikt worden bij de intrastreng basenparen, beschikbaar in de hoofd- en middengroef van de ds-DNA helix (zie Jmol hieronder). Unieke sequenties van basenparen vertonen unieke patronen van H-bindingsdonors en -acceptors in de hoofdgroef. Deze donors/acceptors kunnen worden herkend door specifieke DNA bindende proteïnen die bij binding kunnen leiden tot gen-activering.
- Jmol model van dsDNA met unieke H binding donors en acceptors in de hoofdgroef