DNA
DNA ist ein Polymer, das aus Monomeren, den sogenannten Desoxynukleotiden, besteht. Das Monomer enthält einen einfachen Zucker (Desoxyribose, unten in schwarz dargestellt), eine Phosphatgruppe (in rot) und eine zyklische organische R-Gruppe (in blau), die der Seitenkette einer Aminosäure entspricht.
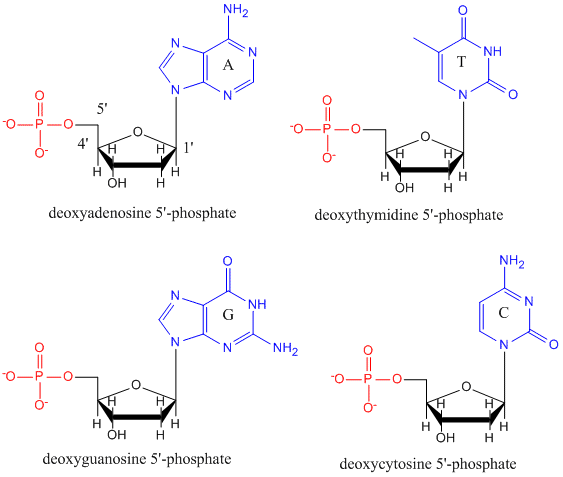
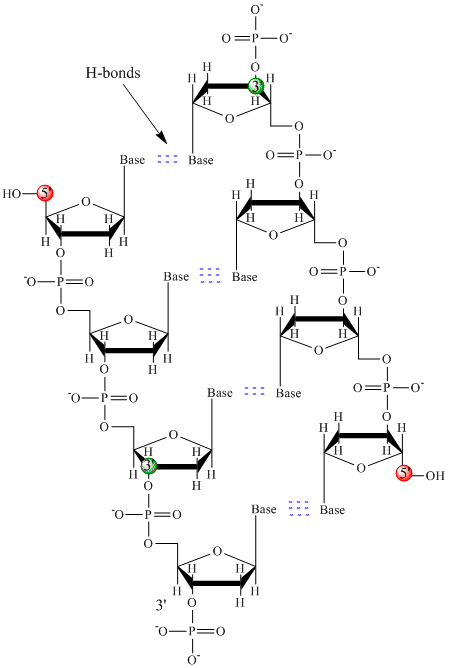
In der DNA werden nur vier Basen verwendet (im Gegensatz zu den 20 verschiedenen Seitenketten in Proteinen), die wir der Einfachheit halber als A, G, C und T abkürzen. Das Polymer besteht aus einem Zucker-Phosphat-Zucker-Phosphat-Grundgerüst, wobei an jedes Zuckermolekül eine Base gebunden ist. Wie bei den Proteinen ist das DNA-Grundgerüst polar, aber auch geladen. Es ist ein Polyanion. Die Basen, die den Seitenketten der Aminosäuren entsprechen, sind überwiegend polar. Angesichts der geladenen Natur des Rückgrats könnte man erwarten, dass sich die DNA nicht zu einer kompakten kugelförmigen Form faltet, auch wenn positiv geladene Kationen wie Mg an das Polymer binden und die Ladung stabilisieren. Stattdessen liegt die DNA normalerweise als Doppelstrang (ds) vor, wobei die Zucker-Phosphat-Rückgrate der beiden Stränge in entgegengesetzter Richtung verlaufen (5′-3′ und der andere 3′-5′). Die Stränge werden durch Wasserstoffbrücken zwischen den Basen auf den komplementären Strängen zusammengehalten. Wie Proteine hat die DNA also eine Sekundärstruktur, aber in diesem Fall befinden sich die Wasserstoffbrücken nicht innerhalb des Rückgrats, sondern zwischen den „Seitenketten“-Basen auf den gegenüberliegenden Strängen. Es ist eigentlich falsch, dsDNA als Molekül zu bezeichnen, da sie in Wirklichkeit aus zwei verschiedenen, komplementären Strängen besteht, die durch Wasserstoffbrücken zusammengehalten werden. Eine Struktur der ds-DNA, die die entgegengesetzte Polarität der Stränge zeigt, ist unten abgebildet.
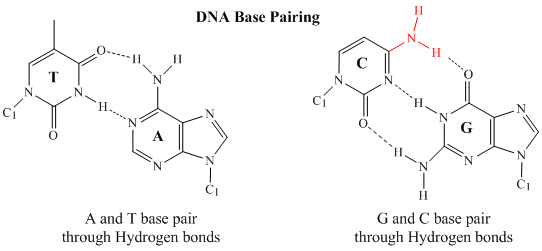
In der doppelsträngigen DNA kann die Guaninbase (G) auf einem Strang drei H-Bindungen mit einer Cytosinbase (C) auf einem anderen Strang eingehen (dies wird als GC-Basenpaar bezeichnet). Die Thyminbase (T) auf einem Strang kann zwei H-Bindungen mit einer Adeninbase (A) auf dem anderen Strang eingehen (dies wird als AT-Basenpaar bezeichnet). Die doppelsträngige DNA hat eine regelmäßige geometrische Struktur mit einem festen Abstand zwischen den beiden Rückgraten. Dies erfordert, dass die Basenpaare aus einer Base mit einer zweiringigen (bicyclischen) Struktur (diese Basen werden Purine genannt) und einer mit einer einringigen Struktur (diese Basen werden Pyrimidine genannt) bestehen. Ein G und A oder ein T und C sind also keine möglichen Basenpaarpartner.
Doppelsträngige DNA variiert in Länge (Anzahl der verbundenen Zucker-Phosphat-Einheiten), Basenzusammensetzung (wie viele von jedem Basensatz) und Sequenz (die Reihenfolge der Basen im Rückgrat). Die folgenden Links bieten interaktive Jmol-Modelle von dsDNA, die von Angel Herráez, Univ. de Alcalá (Spanien) und Eric Martz erstellt wurden.
- Jmol-Modell der ds-DNA mit Basenpaaren und H-Bindungen
- Jmol-Modell der DNA-Stränge und des helikalen Rückgrats
- Jmol-Modell der DNA-Enden und Parallelismen
Chromosomen bestehen aus einer dsDNA mit vielen verschiedenen gebundenen Proteinen. Das menschliche Genom hat etwa 3 Milliarden Basenpaare DNA. Daher hat jedes einzelne Chromosom eines Paares im Durchschnitt etwa 150 Millionen Basenpaare und viele daran gebundene Proteine. dsDNA ist ein stark geladenes Molekül und kann in erster Näherung als ein langes stäbchenförmiges Molekül mit einer großen negativen Ladung betrachtet werden. Es ist ein Polyanion. Dieses sehr große Molekül muss irgendwie in einen kleinen Zellkern einer winzigen Zelle gepackt werden. In komplexen (eukaryotischen) Zellen wird dieses Packungsproblem gelöst, indem die DNA um einen Kernkomplex aus vier verschiedenen Paaren (insgesamt acht Proteine) von Histonproteinen (H2A, H2B, H3 und H4) gewickelt wird, die netto positiv geladen sind. Der Histon-Kernkomplex mit der etwa 2,5-fach gewickelten dsDNA wird als Nukleosom bezeichnet.
Jmol-Modell des Nukleosoms
Die DNA kann zwei andere Arten von Doppelhelixformen annehmen. Die von Watson und Crick entdeckte und in den meisten Lehrbüchern beschriebene Form wird als B-DNA bezeichnet. Je nach der tatsächlichen DNA-Sequenz und dem Hydratationszustand der DNA kann sie dazu gebracht werden, zwei andere Arten von doppelsträngigen Helices zu bilden, nämlich Z- und A-DNA. Die A-Form ist viel offener als die B-Form.
Die 3,2 Milliarden Basenpaare der menschlichen DNA enthalten etwa 24.000 kurze Abschnitte (Gene), die für verschiedene Proteine kodieren. Diese Gene sind in die DNA eingestreut, was mitentscheidend dafür ist, ob das Gen in RNA-Moleküle (siehe unten) und schließlich in Proteine entschlüsselt wird. Damit ein bestimmtes Gen aktiviert (oder „eingeschaltet“) werden kann, müssen bestimmte Proteine an den Bereich eines bestimmten Gens binden. Wie können bindende Proteine spezifische Bindungsziele unter der riesigen Anzahl von Basenpaaren finden, die in erster Näherung eine sich wiederholende Zucker-Phosphat-Basen-Wiederholung aufweisen? Das folgende Jmol zeigt, wie Spezifität erreicht werden kann. Wenn sich die DNA durch Basenpaare zwischen AT und GC zu einer Doppelhelix windet, stehen in der Haupt- und Nebengruppe der ds-DNA-Helix immer noch Wasserstoffbrückenbindungsdonatoren (Amid Hs) und -akzeptoren (Os) an den Basen zur Verfügung, die nicht für die Basenpaarung innerhalb des Strangs verwendet werden (siehe Jmol unten). Einzigartige Basenpaar-Sequenzen weisen einzigartige Muster von H-Bindungsdonatoren und -akzeptoren in der Major Grove auf. Diese Donatoren/Akzeptoren können von spezifischen DNA-Bindungsproteinen erkannt werden, die nach der Bindung zu einer Genaktivierung führen können.
- Jmol-Modell der dsDNA mit einzigartigen H-Bindungsdonatoren und -akzeptoren in der Haupthälfte