DNA
DNA é um polímero, constituído por monómeros chamados de desoxinucleotídeos. O monômero contém um açúcar simples (desoxirribose, mostrado em preto abaixo), um grupo fosfato (em vermelho), e um grupo R cíclico orgânico (em azul) que é análogo à cadeia lateral de um aminoácido.
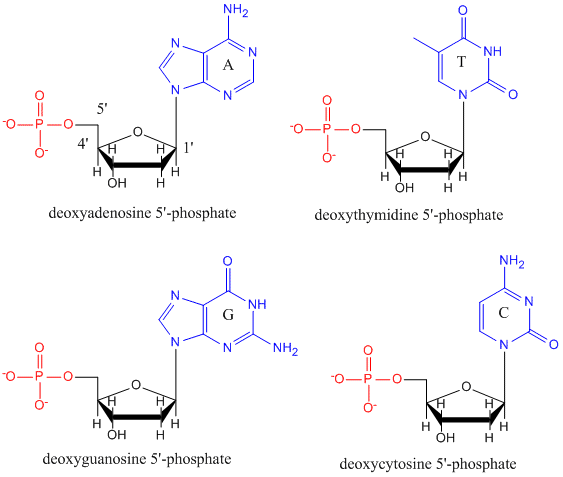
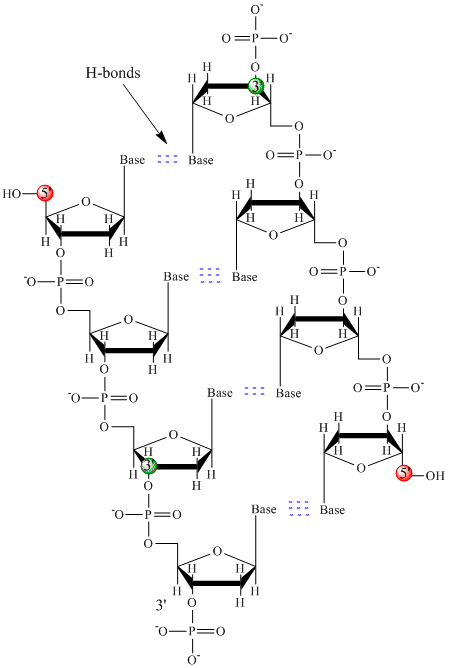
Apenas quatro bases são usadas no DNA (em contraste com as 20 cadeias laterais diferentes em proteínas) que abreviaremos, por simplicidade, como A, G, C e T. São bases já que contêm grupos de aminas que podem aceitar prótons. O polímero consiste em uma espinha dorsal de açúcar – fosfato – açúcar – fosfato, com uma base ligada a cada molécula de açúcar. Tal como as proteínas, a espinha dorsal do ADN é polar, mas também carregada. É um polianion. As bases, análogas às cadeias laterais dos aminoácidos, são predominantemente polares. Dada a natureza carregada da espinha dorsal, você pode esperar que o DNA não dobre para uma forma globular compacta (esférica), mesmo que catiões com carga positiva como Mg se liguem e estabilizem a carga sobre o polímero. Em vez disso, o ADN existe normalmente como uma estrutura de duas cordas (ds) com as espinhas dorsais de fosfato de açúcar das duas cordas diferentes correndo em direcções opostas (5′-3′ e as outras 3′-5′). Os cordões são mantidos juntos por ligações de hidrogênio entre bases sobre cordões complementares. Assim como as proteínas, o DNA tem uma estrutura secundária mas, neste caso, as ligações de hidrogênio não estão dentro da espinha dorsal, mas entre as bases da “cadeia lateral” em filamentos opostos. Na verdade, é um erro de nome chamar dsDNA de molécula, uma vez que na realidade consiste em duas fitas diferentes, complementares, mantidas juntas por ligações de hidrogênio. Uma estrutura de ds-DNA mostrando a polaridade oposta das cordas é mostrada abaixo.
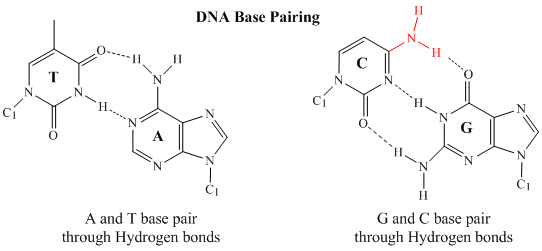
No DNA de cadeia dupla, a base de guanina (G) em uma cordão pode formar três ligações H com uma base de citosina (C) em outro cordão (isto é chamado de par de bases GC). A base timina (T) sobre uma cadeia pode formar dois H-bonds com uma base adenina (A) sobre a outra cadeia (isto é chamado de par de base AT). O DNA de cordão duplo tem uma estrutura geométrica regular com uma distância fixa entre os dois backbones. Isto requer que os pares de bases sejam constituídos por uma base com uma estrutura de dois anéis (bicíclica) (estas bases são chamadas purinas) e uma com uma estrutura de um único anel (estas bases são chamadas pirimidinas). Portanto, um G e A ou um T e C não são possíveis parceiros de pares de bases.
O ADN de dupla cadeia varia em comprimento (número de unidades de fosfato de açúcar ligadas), composição da base (quantas de cada conjunto de bases) e sequência (a ordem das bases na espinha dorsal). Os seguintes links fornecem modelos interativos Jmol de dsDNA feitos por Angel Herráez, Univ. de Alcalá (Espanha) e Eric Martz.
- Modelo Jmol de ds-DNA com pares de bases e ligações H
- Modelo Jmol de fios de ADN e espinha dorsal helicoidal
- Modelo Jmol de pontas de ADN e paralelismos
Cromossomas consistem em um dsDNA com muitas proteínas ligadas diferentes. O genoma humano tem cerca de 3 bilhões de pares de base de DNA. Portanto, em média, cada um dos cromossomas de um par tem cerca de 150 milhões de pares de bases e muitas proteínas ligadas a ele. O dsDNA é uma molécula altamente carregada, e pode ser visto, a uma primeira aproximação, como uma molécula longa semelhante a uma vara com uma grande carga negativa. É um polianion. Esta molécula muito grande deve, de alguma forma, ser acondicionada em um pequeno núcleo de uma célula minúscula. Em células complexas (eucarióticas), este problema de empacotamento é resolvido enrolando o DNA em torno de um núcleo de quatro pares diferentes (oito proteínas no total) de proteínas histônicas (H2A, H2B, H3, e H4) que têm cargas líquidas positivas. O complexo do núcleo histonal com ferida de dsDNA cerca de 2,5 vezes é chamado de nucleossoma.
Jmol modelo do nucleossoma
DNA pode adoptar dois outros tipos de formas de dupla camada. A descoberta por Watson e Crick e encontrada na maioria dos livros didáticos é chamada de B-DNA. Dependendo da seqüência real do DNA e do estado de hidratação do DNA, ele pode ser coaxado para formar dois outros tipos de hélices de dupla camada, o DNA Z e o A. A forma A é muito mais aberta que a forma B.
Os 3,2 bilhões de pares de base de DNA em humanos contêm cerca de 24.000 trechos curtos (genes) que codificam diferentes proteínas. Estes genes são intercalados entre o DNA que ajuda a determinar se o gene é decodificado em moléculas de RNA (veja abaixo) e finalmente em proteínas. Para que um determinado gene seja ativado (ou “ligado”), proteínas específicas devem se ligar à região de um determinado gene. Como as proteínas de ligação podem encontrar alvos de ligação específicos entre o vasto número de pares de bases que, para uma primeira aproximação, têm uma repetição de base de açúcar-fosfato? O Jmol abaixo mostra como a especificidade pode ser alcançada. Quando o DNA se transforma em uma dupla hélice através de pares de bases entre AT e GC, doadores de ligação de hidrogênio (amide Hs) e aceitadores (Os) nas bases que não são usadas no emparelhamento de bases intrastrand, ainda estão disponíveis no maior e menor bosque da hélice ds-DNA (veja Jmol abaixo). Seqüências únicas de pares de bases exibirão padrões únicos de doadores e aceitadores de títulos H no bosque principal. Estes doadores/aceitadores podem ser reconhecidos por proteínas específicas de ligação de DNA que na ligação podem levar à activação do gene.
- Jmol modelo de dsDNA mostrando doadores e aceitadores únicos de ligação de H no bosque maior