DNA
DNA jest polimerem, składającym się z monomerów zwanych deoksynukleotydami. Monomer zawiera cukier prosty (dezoksyrybozę, pokazaną poniżej na czarno), grupę fosforanową (na czerwono) i cykliczną organiczną grupę R (na niebiesko), która jest analogiczna do łańcucha bocznego aminokwasu.
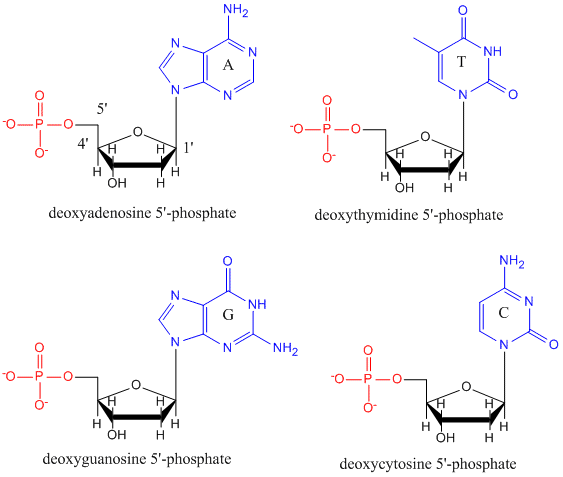
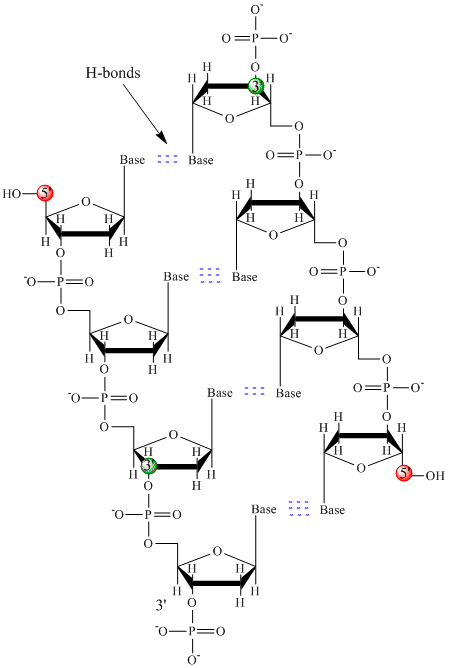
W DNA używane są tylko cztery zasady (w przeciwieństwie do 20 różnych łańcuchów bocznych w białkach), które dla uproszczenia będziemy oznaczać skrótowo jako A, G, C i T. Są one zasadami, ponieważ zawierają grupy aminowe, które mogą przyjmować protony. Polimer składa się z szkieletu cukrowo-fosforanowo-cukrowo-fosforanowego, z jedną zasadą przyłączoną do każdej cząsteczki cukru. Podobnie jak w przypadku białek, szkielet DNA jest polarny, ale również naładowany. Jest to polianion. Zasady, analogicznie do łańcuchów bocznych aminokwasów, są w przeważającej mierze polarne. Biorąc pod uwagę naładowaną naturę szkieletu, można się spodziewać, że DNA nie składa się w zwarty kulisty (sferyczny) kształt, nawet jeśli dodatnio naładowane kationy, takie jak Mg, wiążą się z polimerem i stabilizują jego ładunek. Zamiast tego, DNA istnieje zwykle jako struktura dwuniciowa (ds), w której szkielety cukrowo-fosforanowe dwóch różnych nici biegną w przeciwnych kierunkach (5′-3′ i 3′-5′). Nici są utrzymywane razem przez wiązania wodorowe pomiędzy zasadami na komplementarnych niciach. Stąd, podobnie jak białka, DNA ma strukturę drugorzędową, ale w tym przypadku wiązania wodorowe nie występują w obrębie szkieletu, lecz pomiędzy „łańcuchami bocznymi” zasad na przeciwległych niciach. Nazywanie ds-DNA cząsteczką jest właściwie błędem, ponieważ tak naprawdę składa się ono z dwóch różnych, komplementarnych nici utrzymywanych razem przez wiązania wodorowe. Poniżej przedstawiono strukturę ds-DNA pokazującą przeciwną biegunowość nici.
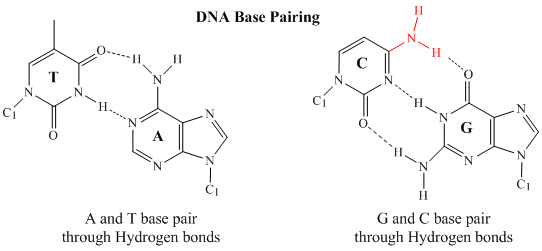
W dwuniciowym DNA, zasada guaninowa (G) na jednej nici może tworzyć trzy wiązania H z zasadą cytozynową (C) na innej nici (jest to nazywane parą zasad GC). Zasada tyminowa (T) na jednej nici może tworzyć dwa wiązania H z zasadą adeninową (A) na drugiej nici (jest to tzw. para zasad AT). Dwuniciowy DNA ma regularną strukturę geometryczną z ustaloną odległością pomiędzy dwoma szkieletami. Wymaga to, aby pary zasad składały się z jednej zasady o strukturze dwupierścieniowej (bicyklicznej) (zasady te nazywane są purynami) i jednej o strukturze jednopierścieniowej (zasady te nazywane są pirymidynami). Stąd G i A lub T i C nie są możliwymi partnerami pary zasad.
Dwuniciowe DNA różni się długością (liczbą połączonych jednostek cukrowo-fosforanowych), składem zasad (ile każdego zestawu zasad) i sekwencją (kolejnością zasad w szkielecie). Poniższe linki zawierają interaktywne modele dsDNA wykonane w języku Jmol przez Angela Herráeza z Univ. de Alcalá (Hiszpania) i Erica Martza.
- Model jmolowy ds-DNA z parami zasad i wiązaniami H
- Model jmolowy nici DNA i spiralnego szkieletu
- Model jmolowy końców DNA i paralelizmów
Chromosomy składają się z jednego dsDNA z wieloma różnymi związanymi białkami. Ludzki genom ma około 3 miliardy par zasad DNA. Dlatego, średnio, każdy pojedynczy chromosom pary ma około 150 milionów par zasad i wiele białek związanych z nim. dsDNA jest silnie naładowaną cząsteczką i może być postrzegany, w pierwszym przybliżeniu, jako długa prętopodobna cząsteczka z dużym ładunkiem ujemnym. Jest to polianion. Ta bardzo duża cząsteczka musi być w jakiś sposób upakowana w małym jądrze maleńkiej komórki. W komórkach złożonych (eukariotycznych) ten problem upakowania jest rozwiązywany przez zwijanie DNA wokół kompleksu rdzeniowego czterech różnych par (łącznie osiem białek) białek histonowych (H2A, H2B, H3 i H4), które mają ładunek dodatni netto. Kompleks rdzenia histonów z dsDNA owiniętym wokół około 2,5 razy nazywany jest nukleosomem.
Jmolowy model nukleosomu
DNA może przyjąć dwa inne rodzaje form dwuhelikalnych. Ten odkryty przez Watsona i Cricka i spotykany w większości podręczników nazywany jest B-DNA. W zależności od rzeczywistej sekwencji DNA i stanu uwodnienia DNA, może on być nakłoniony do utworzenia dwóch innych typów dwuniciowych helikali, Z i A DNA. Forma A jest znacznie bardziej otwarty następnie B form.
The 3,2 miliardów par zasad DNA w ludzi zawiera około 24,000 krótkie odcinki (geny), które kodują różne białka. Te geny są przeplatane wśród DNA, który pomaga określić, czy gen jest dekodowany do cząsteczek RNA (patrz poniżej) i ostatecznie do białek. Aby dany gen został aktywowany (lub „włączony”), specyficzne białka muszą wiązać się z regionem danego genu. W jaki sposób białka wiążące mogą znaleźć specyficzne cele wiązania wśród ogromnej liczby par zasad, które w pierwszym przybliżeniu mają powtarzające się powtórzenia cukrowo-fosforanowo-zasadowe? Jmol poniżej pokazuje, jak można osiągnąć specyficzność. Kiedy DNA zwija się w podwójną helisę poprzez pary zasad między AT i GC, donory wiązań wodorowych (amidowe Hs) i akceptory (Os) na zasadach, które nie są używane w wewnątrzpasmowym parowaniu zasad, są nadal dostępne w głównym i małym rowku helisy ds-DNA (patrz Jmol poniżej). Unikalne sekwencje par zasad będą wykazywać unikalne wzory donorów i akceptorów wiązań H w głównym rowku. Te donory/akceptory mogą być rozpoznawane przez specyficzne białka wiążące DNA, które po związaniu mogą prowadzić do aktywacji genu.
- Jmolowy model dsDNA pokazujący unikalne donory wiązań H i akceptory w głównym rowku