ADN
L’ADN est un polymère, constitué de monomères appelés désoxynucléotides. Le monomère contient un sucre simple (désoxyribose, représenté en noir ci-dessous), un groupe phosphate (en rouge) et un groupe organique cyclique R (en bleu) qui est analogue à la chaîne latérale d’un acide aminé.
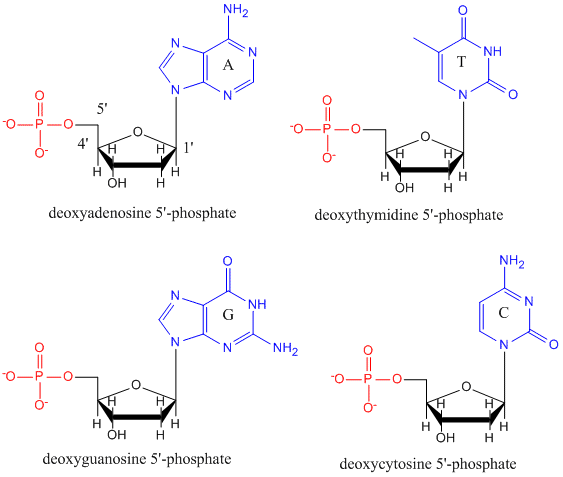
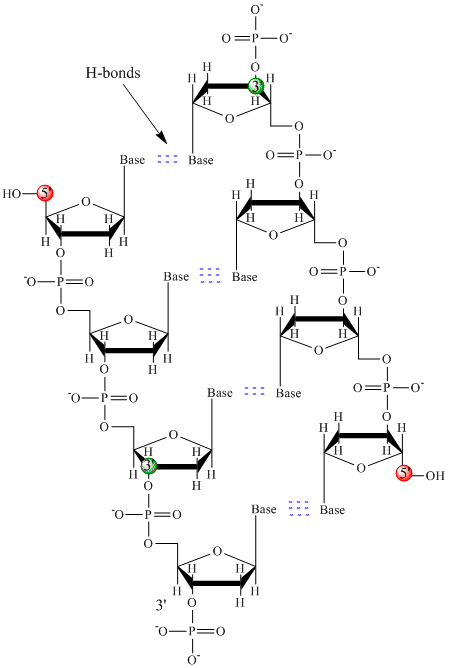
Seulement quatre bases sont utilisées dans l’ADN (contrairement aux 20 chaînes latérales différentes dans les protéines) que nous abrégerons, pour simplifier, par A, G, C et T. Ce sont des bases car elles contiennent des groupes amines qui peuvent accepter des protons. Le polymère est constitué d’un squelette sucre – phosphate – sucre – phosphate, avec une base attachée à chaque molécule de sucre. Comme pour les protéines, le squelette de l’ADN est polaire mais aussi chargé. Il s’agit d’un polyanion. Les bases, analogues aux chaînes latérales des acides aminés, sont principalement polaires. Étant donné la nature chargée du squelette, on peut s’attendre à ce que l’ADN ne se plie pas en une forme globulaire compacte (sphérique), même si des cations chargés positivement comme le magnésium se lient au polymère et en stabilisent la charge. Au lieu de cela, l’ADN existe généralement sous la forme d’une structure à double brin (ds), les squelettes de sucre-phosphate des deux brins différents étant orientés dans des directions opposées (5′-3′ et l’autre 3′-5′). Les brins sont maintenus ensemble par des liaisons hydrogène entre les bases des brins complémentaires. Ainsi, comme les protéines, l’ADN possède une structure secondaire, mais dans ce cas, les liaisons hydrogène ne se situent pas à l’intérieur du squelette, mais entre les bases de la « chaîne latérale » sur les brins opposés. Il est en fait erroné d’appeler l’ADNdb une molécule, car il s’agit en réalité de deux brins différents et complémentaires maintenus ensemble par des liaisons hydrogène. Une structure d’ADNdb montrant la polarité opposée des brins est illustrée ci-dessous.
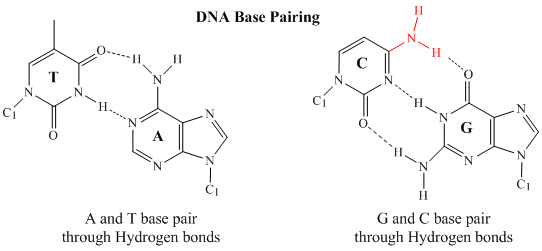
Dans l’ADN double brin, la base guanine (G) sur un brin peut former trois liaisons H avec une base cytosine (C) sur un autre brin (c’est ce qu’on appelle une paire de bases GC). La base thymine (T) d’un brin peut former deux liaisons H avec une base adénine (A) de l’autre brin (c’est ce qu’on appelle une paire de bases AT). L’ADN double brin a une structure géométrique régulière avec une distance fixe entre les deux squelettes. Les paires de bases doivent donc être constituées d’une base ayant une structure à deux cycles (bicyclique) (ces bases sont appelées purines) et d’une base ayant une structure à un seul cycle (ces bases sont appelées pyrimidines). Par conséquent, un G et un A ou un T et un C ne sont pas des partenaires de paires de bases possibles.
L’ADN double brin varie en longueur (nombre d’unités sucre-phosphate connectées), en composition des bases (combien de chaque ensemble de bases) et en séquence (l’ordre des bases dans le squelette). Les liens suivants fournissent des modèles Jmol interactifs d’ADNdb réalisés par Angel Herráez, Univ. de Alcalá (Espagne) et Eric Martz.
- Modèle Jmol d’ADNdb avec paires de bases et liaisons H
- Modèle Jmol de brins d’ADN et squelette hélicoïdal
- Modèle Jmol d’extrémités d’ADN et parallélismes
Les chromosomes sont constitués d’un ADNdb avec de nombreuses protéines différentes liées. Le génome humain compte environ 3 milliards de paires de bases d’ADN. Par conséquent, en moyenne, chaque chromosome unique d’une paire a environ 150 millions de paires de bases et beaucoup de protéines liées à lui. L’ADNdb est une molécule hautement chargée, et peut être vu, en première approximation, comme une longue molécule en forme de tige avec une grande charge négative. Il s’agit d’un polyanion. Cette très grande molécule doit, d’une manière ou d’une autre, être emballée dans le petit noyau d’une minuscule cellule. Dans les cellules complexes (eucaryotes), ce problème d’entassement est résolu en enroulant l’ADN autour d’un complexe central composé de quatre paires différentes (huit protéines au total) de protéines histones (H2A, H2B, H3 et H4) qui ont des charges positives nettes. Le complexe central d’histone avec l’ADNdb enroulé environ 2,5 fois est appelé le nucléosome.
Modèle Jmol du nucléosome
L’ADN peut adopter deux autres types de formes à double hélice. Celle qui a été découverte par Watson et Crick et que l’on retrouve dans la plupart des manuels est appelée ADN B. Selon la séquence réelle de l’ADN et l’état d’hydratation de l’ADN, il peut être amené à former deux autres types d’hélices à double brin, l’ADN Z et l’ADN A. La forme A est beaucoup plus ouverte que la forme B.
Les 3,2 milliards de paires de bases d’ADN chez les humains contiennent environ 24 000 courts tronçons (gènes) qui codent pour différentes protéines. Ces gènes sont intercalés dans l’ADN qui aide à déterminer si le gène est décodé en molécules d’ARN (voir ci-dessous) et finalement en protéines. Pour qu’un gène particulier soit activé (ou « allumé »), des protéines spécifiques doivent se lier à la région d’un gène particulier. Comment les protéines de liaison peuvent-elles trouver des cibles de liaison spécifiques parmi le grand nombre de paires de bases qui, en première approximation, ont une répétition sucre-phosphate-base répétitive ? Le Jmol ci-dessous montre comment la spécificité peut être atteinte. Lorsque l’ADN s’enroule en une double hélice par le biais de paires de bases entre AT et GC, des donneurs de liaisons hydrogène (Hs amides) et des accepteurs (Os) sur les bases qui ne sont pas utilisées dans l’appariement de bases intrastrand, sont encore disponibles dans le sillon majeur et mineur de l’hélice d’ADNd (voir Jmol ci-dessous). Les séquences de paires de bases uniques présenteront des modèles uniques de donneurs et d’accepteurs de liaisons H dans le sillon majeur. Ces donneurs/accepteurs peuvent être reconnus par des protéines spécifiques de liaison à l’ADN qui, lors de la liaison, peuvent conduire à l’activation du gène.
- Modèle Jmol d’ADNd montrant des donneurs et des accepteurs de liaison H uniques dans le grand sillon
.