DNA
Il DNA è un polimero, costituito da monomeri chiamati deossinucleotidi. Il monomero contiene uno zucchero semplice (desossiribosio, mostrato in nero sotto), un gruppo fosfato (in rosso), e un gruppo organico ciclico R (in blu) che è analogo alla catena laterale di un aminoacido.
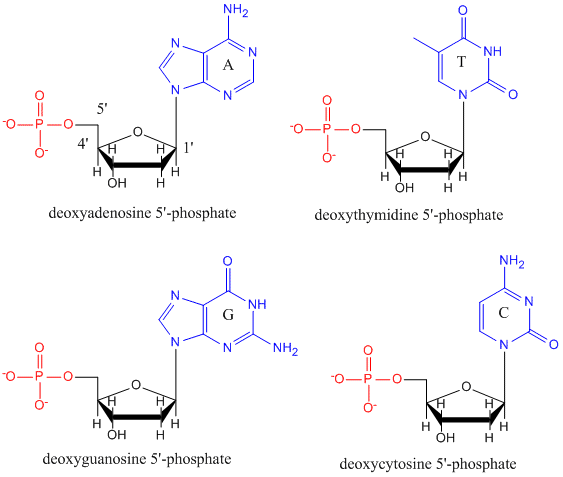
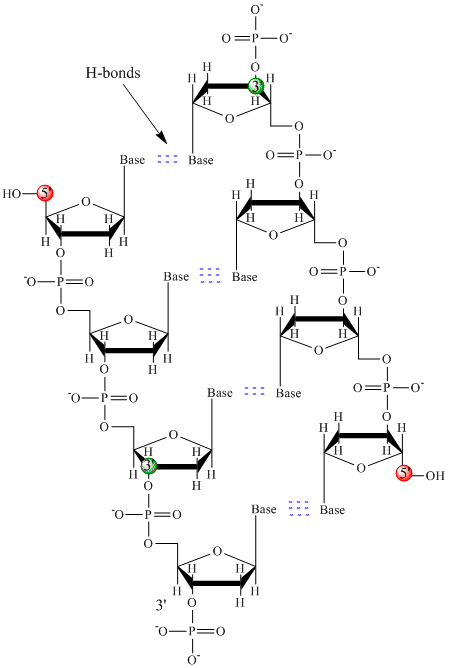
Nel DNA si usano solo quattro basi (in contrasto con le 20 diverse catene laterali delle proteine) che abbrevieremo, per semplicità, come A, G, C e T. Sono basi perché contengono gruppi amminici che possono accettare protoni. Il polimero consiste in una spina dorsale zucchero – fosfato – zucchero – fosfato, con una base attaccata ad ogni molecola di zucchero. Come per le proteine, la spina dorsale del DNA è polare ma anche carica. È un polianione. Le basi, analogamente alle catene laterali degli aminoacidi, sono prevalentemente polari. Data la natura carica della spina dorsale, ci si potrebbe aspettare che il DNA non si ripieghi in una forma globulare compatta (sferica), anche se cationi con carica positiva come il Mg si legano e stabilizzano la carica sul polimero. Invece, il DNA esiste di solito come una struttura a doppio filamento (ds) con le dorsali di zucchero-fosfato dei due diversi filamenti che corrono in direzioni opposte (5′-3′ e l’altro 3′-5′). I filamenti sono tenuti insieme da legami idrogeno tra le basi sui filamenti complementari. Quindi, come le proteine, il DNA ha una struttura secondaria, ma in questo caso, i legami a idrogeno non sono all’interno della spina dorsale, ma tra le basi della “catena laterale” sui filamenti opposti. In realtà è un termine improprio chiamare il dsDNA una molecola, poiché in realtà consiste di due diversi filamenti complementari tenuti insieme da legami a idrogeno. Una struttura del ds-DNA che mostra la polarità opposta dei filamenti è mostrata qui sotto.
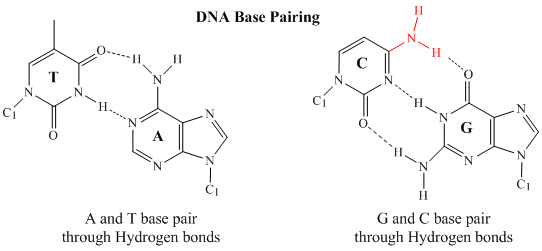
Nel DNA a doppio filamento, la base guanina (G) su un filamento può formare tre legami H con una base citosina (C) su un altro filamento (questa è chiamata una coppia di basi GC). La base timina (T) su un filamento può formare due legami H con una base adenina (A) sull’altro filamento (questa è chiamata una coppia di basi AT). Il DNA a doppio filamento ha una struttura geometrica regolare con una distanza fissa tra i due backbone. Ciò richiede che le coppie di basi siano costituite da una base con una struttura a due anelli (biciclica) (queste basi sono chiamate purine) e una con una struttura ad anello singolo (queste basi sono chiamate pirimidine). Quindi una G e una A o una T e una C non sono possibili partner di coppia di basi.
Il DNA a doppio filamento varia in lunghezza (numero di unità di zucchero-fosfato collegate), composizione delle basi (quante di ciascuna serie di basi) e sequenza (l’ordine delle basi nella spina dorsale). I seguenti link forniscono modelli Jmol interattivi di dsDNA fatti da Angel Herráez, Univ. de Alcalá (Spagna) e Eric Martz.
- Modello Jmol di ds-DNA con coppie di basi e legami H
- Modello Jmol di filamenti di DNA e spina dorsale elicoidale
- Modello Jmol di estremità di DNA e parallelismi
I cromosomi consistono in un dsDNA con molte diverse proteine legate. Il genoma umano ha circa 3 miliardi di paia di basi di DNA. Quindi, in media, ogni singolo cromosoma di una coppia ha circa 150 milioni di coppie di basi e molte proteine legate ad esso. Il dsDNA è una molecola altamente carica, e può essere visto, in prima approssimazione, come una lunga molecola a forma di asta con una grande carica negativa. È un polianione. Questa molecola molto grande deve in qualche modo essere impacchettata in un piccolo nucleo di una minuscola cellula. Nelle cellule complesse (eucariotiche), questo problema di impacchettamento è risolto avvolgendo il DNA attorno a un complesso centrale di quattro diverse coppie (otto proteine in totale) di proteine istone (H2A, H2B, H3 e H4) che hanno cariche positive nette. Il complesso del nucleo istonico con il dsDNA avvolto circa 2,5 volte è chiamato nucleosoma.
Modello Jmol del nucleosoma
Il DNA può adottare altri due tipi di forme a doppia elica. Quella scoperta da Watson e Crick e presente nella maggior parte dei libri di testo è chiamata B-DNA. A seconda della sequenza attuale del DNA e dello stato di idratazione del DNA, esso può essere spinto a formare altri due tipi di eliche a doppio filamento, Z e A DNA. La forma A è molto più aperta della forma B.
I 3,2 miliardi di paia di basi del DNA negli esseri umani contengono circa 24.000 brevi tratti (geni) che codificano diverse proteine. Questi geni sono intervallati da DNA che aiuta a determinare se il gene è decodificato in molecole di RNA (vedi sotto) e infine in proteine. Perché un particolare gene sia attivato (o “acceso”), proteine specifiche devono legarsi alla regione di un particolare gene. Come possono le proteine leganti trovare bersagli specifici tra il vasto numero di coppie di basi che, in prima approssimazione, hanno una ripetizione zucchero-fosfato-base? Il Jmol qui sotto mostra come si può ottenere la specificità. Quando il DNA si avvolge in una doppia elica attraverso le coppie di basi tra AT e GC, i donatori di legami a idrogeno (ammide Hs) e gli accettatori (Os) sulle basi che non sono usati nell’accoppiamento di basi intrastrand, sono ancora disponibili nel boschetto maggiore e minore dell’elica del ds-DNA (vedi Jmol sotto). Sequenze uniche di coppie di basi mostreranno modelli unici di donatori e accettatori di legami H nel solco maggiore. Questi donatori/accettori possono essere riconosciuti da specifiche proteine leganti il DNA che, legandosi, possono portare all’attivazione del gene.
- Modello Jmol di dsDNA che mostra donatori e accettatori di legami H unici nel solco maggiore