DNA
DNA är en polymer som består av monomerer som kallas desoxynukleotider. Monomeren innehåller ett enkelt socker (deoxyribose, visas i svart nedan), en fosfatgrupp (i rött) och en cyklisk organisk R-grupp (i blått) som är analog med sidokedjan i en aminosyra.
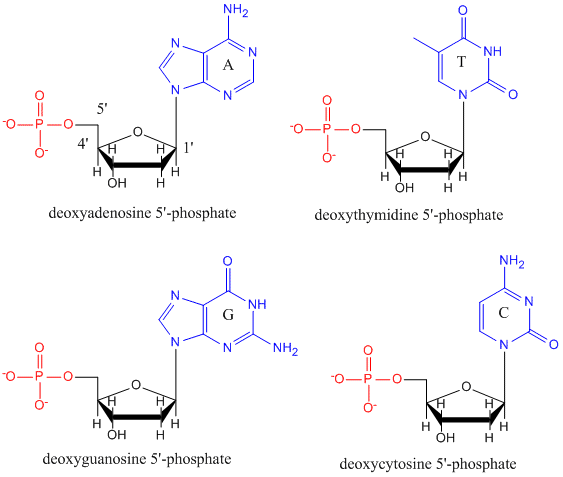
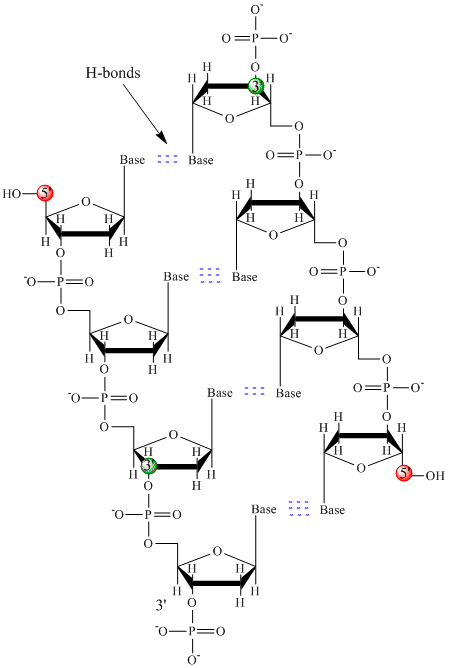
Endast fyra baser används i DNA (till skillnad från de 20 olika sidokedjorna i proteiner) som vi för enkelhetens skull förkortar med A, G, C och T. De är baser eftersom de innehåller amingrupper som kan ta emot protoner. Polymeren består av en ryggrad av socker – fosfat – socker – fosfat, med en bas knuten till varje sockermolekyl. Precis som för proteiner är DNA-ryggen polär men också laddad. Det är en polyanjon. Baserna, som motsvarar aminosyrornas sidokedjor, är huvudsakligen polära. Med tanke på ryggradets laddade natur kan man förvänta sig att DNA inte viks till en kompakt globulär (sfärisk) form, även om positivt laddade katjoner som Mg binder till och stabiliserar laddningen på polymeren. Istället existerar DNA vanligtvis som en dubbelsträngad (ds) struktur där sockerfosfatryggarna i de två olika strängarna löper i motsatt riktning (5′-3′ och den andra 3′-5′). Strängarna hålls ihop av vätebindningar mellan baser på komplementära strängar. Precis som proteiner har DNA alltså en sekundär struktur, men i det här fallet finns vätebindningarna inte inom ryggraden utan mellan baserna med ”sidokedjor” på motsatta strängar. Det är faktiskt en felaktig benämning att kalla dsDNA för en molekyl, eftersom det i själva verket består av två olika, komplementära strängar som hålls samman av vätebindningar. En struktur av ds-DNA som visar strängarnas motsatta polaritet visas nedan.
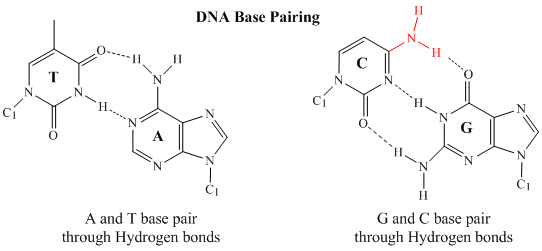
I dubbelsträngat DNA kan guaninbasen (G) på en sträng bilda tre H-bindningar med en cytosinbas (C) på en annan sträng (detta kallas ett GC-baspar). Tyminbasen (T) på den ena strängen kan bilda två H-bindningar med en adeninbas (A) på den andra strängen (detta kallas ett AT-baspar). Dubbelsträngat DNA har en regelbunden geometrisk struktur med ett fast avstånd mellan de två ryggraden. Detta kräver att basparen består av en bas med en tvåringsstruktur (bicyklisk) (dessa baser kallas puriner) och en bas med en enkelringsstruktur (dessa baser kallas pyrimidiner). Därför är ett G och A eller ett T och C inte möjliga basparpartners.
Dubbelsträngat DNA varierar i längd (antal kopplade socker-fosfat-enheter), bassammansättning (hur många av varje uppsättning baser) och sekvens (ordningen på baserna i ryggraden). Följande länkar innehåller interaktiva Jmol-modeller av dsDNA som skapats av Angel Herráez, Univ. de Alcalá (Spanien) och Eric Martz.
- Jmol-modell av ds-DNA med baspar och H-bindningar
- Jmol-modell av DNA-strängar och spiralformad ryggrad
- Jmol-modell av DNA-ändar och parallellism
Chromosomer består av ett dsDNA med många olika bundna proteiner. Det mänskliga genomet består av cirka 3 miljarder baspar av dna. Därför har i genomsnitt varje enskild kromosom i ett par ungefär 150 miljoner baspar och massor av proteiner bundna till sig. dsDNA är en starkt laddad molekyl och kan i en första approximation ses som en lång stavliknande molekyl med en stor negativ laddning. Det är en polyanjon. Denna mycket stora molekyl måste på något sätt packas in i en liten cellkärna i en liten cell. I komplexa (eukaryota) celler löses detta packningsproblem genom att DNA lindas runt ett kärnkomplex av fyra olika par (totalt åtta proteiner) av histonproteiner (H2A, H2B, H3 och H4) som har positiva nettoladdningar. Histonkärnkomplexet med dsDNA lindat runt ungefär 2,5 gånger kallas nukleosomen.
Jmolmodell av nukleosomen
DNA kan anta två andra typer av dubbelhelikala former. Den som upptäcktes av Watson och Crick och som finns med i de flesta läroböcker kallas B-DNA. Beroende på den faktiska DNA-sekvensen och DNA:s hydreringstillstånd kan det övertalas att bilda två andra typer av dubbelsträngade helikser, Z- och A-DNA. A-formen är mycket öppnare än B-formen.
De 3,2 miljarder baspar DNA i människan innehåller cirka 24 000 korta sträckor (gener) som kodar för olika proteiner. Dessa gener är interfolierade bland DNA som hjälper till att avgöra om genen avkodas till RNA-molekyler (se nedan) och i slutändan till proteiner. För att en viss gen ska aktiveras (eller ”slås på”) måste specifika proteiner binda till området i en viss gen. Hur kan bindande proteiner hitta specifika bindningsmål bland det stora antalet baspar som i första hand har en repetitiv upprepning av sockerfosfatbasen? Jmol nedan visar hur specificitet kan uppnås. När DNA slingrar sig till en dubbelspiral genom baspar mellan AT och GC, finns det fortfarande vätebindningsdonatorer (amid Hs) och acceptorer (Os) på de baser som inte används vid basparning inom strängen, tillgängliga i ds-DNA-helixens stora och lilla lund (se Jmol nedan). Unika basparsekvenser kommer att uppvisa unika mönster av givare och mottagare av H-bindningar i major grove. Dessa donatorer/acceptorer kan kännas igen av specifika DNA-bindningsproteiner som vid bindning kan leda till genaktivering.
- Jmol-modell av dsDNA som visar unika H-bindningsdonatorer och acceptorer i major grove